Ligation sequencing V14 - PCR barcoding (SQK-LSK114 with EXP-PBC001 or EXP-PBC096) (PBC_9182_v114_revP_24Sep2025)
MinION: Protocol
V PBC_9182_v114_revP_24Sep2025
FOR RESEARCH USE ONLY
Contents
Introduction to the protocol
Library preparation
- 3. End-prep
- 4. Ligation of Barcode Adapter
- 5. Barcoding PCR
- 6. End-prep
- 7. Adapter ligation and clean-up
- 8. Priming and loading the MinION and GridION Flow Cell
Sequencing and data analysis
Troubleshooting
1. Overview of the protocol
PCR Barcoding Expansion features:
We have two PCR barcoding expansions available depending on the number of barcodes required:
- PCR Barcoding Expansion 1-12 (EXP-PBC001): up to 12 unique barcodes are available.
- PCR Barcoding Expansion 1-96 (EXP-PBC096): up to 96 unique barcodes are available.
These expansions are used with the Ligation Sequencing Kit V14 and recommended for:
- Multiplexing up to 12 or 96 barcodes, depending on the expansion you are using.
- Achieving raw read sequencing modal accuracy of Q20+ (99%) or above.
- Optimising your sequencing experiment for output.
- Control over read length.
- Utilising upstream processes such as size selection or whole genome amplification.
Introduction to the PCR Barcoding protocol
This protocol describes how to carry out PCR barcoding of gDNA using the Ligation Sequencing Kit V14 (SQK-LSK114) with the PCR Barcoding Expansion Pack 1-12 (EXP-PBC001) or 1-96 (EXP-PBC096). It also outlines recommendations to PCR barcode amplicons. Using the PCR Barcoding expansions allow for up to either 12 or 96 samples to be combined and loaded onto a single flow cell. It is highly recommended that a Lambda control experiment is completed first to become familiar with the technology.
Note: For amplicon inputs, first-round PCR product with the following tailed primers are required. 5’ TTTCTGTTGGTGCTGATATTGC-[ project-specific forward primer sequence ] 3’ 5’ ACTTGCCTGTCGCTCTATCTTC-[ project-specific reverse primer sequence ] 3’
Steps in the sequencing workflow:
Prepare for your experiment You will need to:
- Extract your DNA, and check its length, quantity and purity. The quality checks performed during the protocol are essential in ensuring experimental success.
- Ensure you have your sequencing kit, the correct equipment and third-party reagents.
- Download the software for acquiring and analysing your data.
- Check your flow cell to ensure it has enough pores for a good sequencing run.
Experiment workflow
The Table below is an overview of the steps required in the sample and library preparation, including timings and optional stopping points.
| Library preparation | Process | Time | Stop option |
|---|---|---|---|
| For gDNA: DNA end-prep or For amplicons: Incorporate tailed primers | Prepare the DNA ends for PCR adapter attachment or Perform a round of PCR to incoporate tailed primers | 35 minutes | 4°C overnight |
| For gDNA: attach barcoding adapters or For amplicons: second round of PCR to incorporate barcode sequences | Attach barcoding adapters to the DNA ends. or Complete a second round of PCR to incorporate the Oxford Nanopore barcode sequences. | 40 minutes | 4°C overnight |
| Amplify and pool samples | Amplify each barcoded sample by PCR, then pool the samples together. | 15 minutes + Adjustable PCR time | 4°C overnight |
| End-prep | Repair the DNA and prepare the DNA ends for adapter attachment | 35 minutes | 4°C overnight |
| Adapter ligation and clean-up | Attach the sequencing adapters to the DNA ends | 20 minutes | 4°C short-term storage or for repeated use, such as re-loading your flow cell -80°C for single-use, long-term storage. We strongly recommend sequencing your library as soon as it is adapted. |
| Priming and loading the flow cell | Prime the flow cell and load the prepared library for sequencing | 10 minutes |
Sequencing and analysis
You will need to:
- Select the Ligation Sequencing Kit and PCR Barcoding Expansion in "Kit selection" when setting up a sequencing run in the current version of the MinKNOW software.
- Start a sequencing run in the MinKNOW software, which will collect raw data from the device and convert it into basecalled reads.
- Start the EPI2ME software and select a workflow for further analysis (this step is optional).
We do not recommend mixing barcoded libraries with non-barcoded libraries prior to sequencing.
Compatibility of this protocol
This protocol should only be used in combination with:
- PCR Barcoding Expansion Pack 1-12 (EXP-PBC001)
- PCR Barcoding Expansion Pack 1-96 (EXP-PBC096)
- Ligation Sequencing Kit V14 (SQK-LSK114)
- R10.4.1 MinION Flow Cells (FLO-MIN114)
- Flow Cell Wash Kit (EXP-WSH004)
- Control Expansion (EXP-CTL001)
- Flow Cell Priming Kit V14 (EXP-FLP004)
- MinION Mk1D - MinION Mk1D IT requirements document
- GridION - GridION IT requirements document
2. Equipment and consumables
材料
- 100 ng of each sheared DNA sample to be barcoded in 45 µl
- OR 100 ng first-round PCR product (with tailed primers) per sample
- PCR Barcoding Expansion 1-12 (EXP-PBC001)
- PCR Barcoding Expansion 1-96 (EXP-PBC096)
- 连接测序试剂盒V14(SQK-LSK114)
耗材
- MinION/GridION测序芯片
- LongAmp 热启动 Taq 酶 2X 预混液(NEB, M0533)
- NEBNext Ultra II 末端修复/ dA尾添加模块(NEB,E7546)
- 耐盐T4 DNA连接酶(NEB, M0467)
- NEB Blunt/TA 连接酶预混液(NEB,M0367)
- Agencourt AMPure XP beads (Beckman Coulter, A63881)
- 1.5 ml Eppendorf DNA LoBind 离心管
- 0.2 ml 薄壁 PCR 离心管 或 0.2 ml 96 孔 PCR 板
- 新制备的 80% 乙醇(用无核酸酶水配制)
- 无核酸酶水(如ThermoFisher,AM9937)
- Qubit™ 分析管(Invitrogen, Q32856)
- Qubit dsDNA HS Assay(双链DNA高灵敏度检测)试剂盒(Invitrogen, Q32851)
- 牛血清白蛋白(BSA)(50 mg/mL)(例如 Invitrogen™ UltraPure™ BSA 50 mg/mL, AM2616)
仪器
- MinION 或 GridION 测序仪
- MinION 及GridION 测序芯片遮光片
- Hula混匀仪(低速旋转式混匀仪)
- 迷你离心机
- 涡旋混匀仪
- 热循环仪
- P1000 移液枪和枪头
- P200 移液枪和枪头
- P100 移液枪和枪头
- P20 移液枪和枪头
- P10 移液枪和枪头
- P2 移液枪和枪头
- 盛有冰的冰桶
- 磁力架
- 计时器
- 多通道移液枪和枪头
- Qubit™ 荧光计(或用于质控检测的等效仪器)
可选仪器
- Agilent 生物分析仪(或等效仪器)
- Eppendorf 5424 离心机(或等效器材)
100 ng of gDNA is required per sample.
Optional: If using amplicons, 100 ng of first-round PCR product (with tailed primers) is required per sample.
Start from the "Barcoding PCR" step after the amplicons have undergone a first round of PCR to incorporate the following 5' tail sequences:
- 5’ TTTCTGTTGGTGCTGATATTGC-[ project-specific forward primer sequence ] 3’
- 5’ ACTTGCCTGTCGCTCTATCTTC-[ project-specific reverse primer sequence ] 3’
The sequences above are required for the Barcoding PCR step to incorporate the Oxford Nanopore barcode sequences into your amplicon. Multiple first-round PCR products can be pooled together and we recommend all amplicon samples receiving the same barcode should be quantified and pooled in the desired ratios before the barcoding PCR step is performed.
After the first round of PCR to incorporate the 5' tail is complete, a purification step is required to remove any proteins, salts, dNTPs and primers. We recommend using AMPure XP Beads but other methods suitable for the amplicon size may also be used.
If you do not want to redesign your primers, the Barcode Adapter (BCA) from the PCR Barcoding Expansion 1-12 or 1-96 (EXP-PBC001 or EXP-PBC096) can be ligated onto the ends of the amplicons and the protocol started from the first end-prep step.
Fragmentation and size selection
For successful amplification, it is critical that the DNA fragment distribution of templates used in the PCR are <8 kb. If the input DNA is >8 kb, follow the steps outlined in Shearing genomic DNA using the Covaris g-TUBE™ to shear the sample so that fragment distribution is <8 kb.
Additionally, we offer several options for size-selecting your DNA sample to enrich for long fragments. Instructions are available in the Size Selection section of Extraction methods.
Input DNA
How to QC your input DNA
It is important that the input DNA meets the quantity and quality requirements. Using too little or too much DNA, or DNA of poor quality (e.g. highly fragmented or containing RNA or chemical contaminants) can affect your library preparation.
For instructions on how to perform quality control of your DNA sample, please read the Input DNA/RNA QC protocol.
Chemical contaminants
Depending on how the DNA is extracted from the raw sample, certain chemical contaminants may remain in the purified DNA, which can affect library preparation efficiency and sequencing quality. Read more about contaminants on the Contaminants page of the Community.
Third-party reagents
We have validated and recommend the use of all the third-party reagents used in this protocol. Alternatives have not been tested by Oxford Nanopore Technologies.
For all third-party reagents, we recommend following the manufacturer's instructions to prepare the reagents for use.
Check your flow cell
We highly recommend that you check the number of pores in your flow cell prior to starting a sequencing experiment. This should be done within 12 weeks of purchasing your MinION/GridION/PromethION Flow Cells. Oxford Nanopore Technologies will replace any unused flow cell with fewer than the number of pores listed in the Table below, when the result is reported within two days of performing the flow cell check, and when the storage recommendations have been followed. To do the flow cell check, please follow the instructions in the Flow Cell Check document.
| Flow cell | Minimum number of active pores covered by warranty |
|---|---|
| MinION/GridION Flow Cell | 800 |
| PromethION Flow Cell | 5000 |
AMPure XP Beads
Within the Ligation Sequencing Kit V14 (SQK-LSK114), AMPure XP Beads (AXP) are supplied at the volume needed to complete the "End-prep" and "Adapter ligation and clean up" steps of the protocol. However, for initially preparing the sample ends and the PCR steps, extra AMPure XP Beads are required for purification and clean-up. Please note, other purification methods are available.
Ligation Sequencing Kit V14 (SQK-LSK114) contents
Note: This product contains AMPure XP reagent manufactured by Beckman Coulter, Inc. and can be stored at -20°C with the kit without detriment to reagent stability.
Note: The DNA Control Sample (DCS) is a 3.6 kb standard amplicon mapping the 3' end of the Lambda genome.
PCR Barcoding Expansion 1-12 (EXP-PBC001) contents
| Name | Acronym | Cap colour | No. of vials | Fill volume per vial (μl) |
|---|---|---|---|---|
| PCR Barcode 1-12 | BC1-12 | Clear | 12 | 20 |
| Barcode Adapter | BCA | Blue stripe | 6 | 260 |
PCR Barcoding Expansion Pack 1-96 (EXP-PBC096)
Layout of barcodes in the 96 tube plate
The wells of the 96 tube plate correspond to the barcodes in the following way. All barcodes are supplied at 10 µM concentration and to be used at a final concentration of 0.2 µM.
96 barcode sequences
| Component | Sequence |
|---|---|
| BC01 / RB01 | AAGAAAGTTGTCGGTGTCTTTGTG |
| BC02 / RB02 | TCGATTCCGTTTGTAGTCGTCTGT |
| BC03 / RB03 | GAGTCTTGTGTCCCAGTTACCAGG |
| BC04 / RB04 | TTCGGATTCTATCGTGTTTCCCTA |
| BC05 / RB05 | CTTGTCCAGGGTTTGTGTAACCTT |
| BC06 / RB06 | TTCTCGCAAAGGCAGAAAGTAGTC |
| BC07 / RB07 | GTGTTACCGTGGGAATGAATCCTT |
| BC08 / RB08 | TTCAGGGAACAAACCAAGTTACGT |
| BC09 / RB09 | AACTAGGCACAGCGAGTCTTGGTT |
| BC10 / RB10 | AAGCGTTGAAACCTTTGTCCTCTC |
| BC11 / RB11 | GTTTCATCTATCGGAGGGAATGGA |
| BC12 / RB12 | CAGGTAGAAAGAAGCAGAATCGGA |
| BC13 / 16S13 / RB13 | AGAACGACTTCCATACTCGTGTGA |
| BC14 / 16S14 / RB14 | AACGAGTCTCTTGGGACCCATAGA |
| BC15 / 16S15 / RB15 | AGGTCTACCTCGCTAACACCACTG |
| BC16 / 16S16 / RB16 | CGTCAACTGACAGTGGTTCGTACT |
| BC17 / 16S17 / RB17 | ACCCTCCAGGAAAGTACCTCTGAT |
| BC18 / 16S18 / RB18 | CCAAACCCAACAACCTAGATAGGC |
| BC19 / 16S19 / RB19 | GTTCCTCGTGCAGTGTCAAGAGAT |
| BC20 / 16S20 / RB20 | TTGCGTCCTGTTACGAGAACTCAT |
| BC21 / 16S21 / RB21 | GAGCCTCTCATTGTCCGTTCTCTA |
| BC22 / 16S22 / RB22 | ACCACTGCCATGTATCAAAGTACG |
| BC23 / 16S23 / RB23 | CTTACTACCCAGTGAACCTCCTCG |
| BC24 / 16S24 / RB24 | GCATAGTTCTGCATGATGGGTTAG |
| BC25 / RB25 | GTAAGTTGGGTATGCAACGCAATG |
| BC26 / RB26 | CATACAGCGACTACGCATTCTCAT |
| BC27 / RB27 | CGACGGTTAGATTCACCTCTTACA |
| BC28 / RB28 | TGAAACCTAAGAAGGCACCGTATC |
| BC29 / RB29 | CTAGACACCTTGGGTTGACAGACC |
| BC30 / RB30 | TCAGTGAGGATCTACTTCGACCCA |
| BC31 / RB31 | TGCGTACAGCAATCAGTTACATTG |
| BC32 / RB32 | CCAGTAGAAGTCCGACAACGTCAT |
| BC33 / RB33 | CAGACTTGGTACGGTTGGGTAACT |
| BC34 / RB34 | GGACGAAGAACTCAAGTCAAAGGC |
| BC35 / RB35 | CTACTTACGAAGCTGAGGGACTGC |
| BC36 / RB36 | ATGTCCCAGTTAGAGGAGGAAACA |
| BC37 / RB37 | GCTTGCGATTGATGCTTAGTATCA |
| BC38 / RB38 | ACCACAGGAGGACGATACAGAGAA |
| BC39 / RB39 | CCACAGTGTCAACTAGAGCCTCTC |
| BC40 / RB40 | TAGTTTGGATGACCAAGGATAGCC |
| BC41 / RB41 | GGAGTTCGTCCAGAGAAGTACACG |
| BC42 / RB42 | CTACGTGTAAGGCATACCTGCCAG |
| BC43 / RB43 | CTTTCGTTGTTGACTCGACGGTAG |
| BC44 / RB44 | AGTAGAAAGGGTTCCTTCCCACTC |
| BC45 / RB45 | GATCCAACAGAGATGCCTTCAGTG |
| BC46 / RB46 | GCTGTGTTCCACTTCATTCTCCTG |
| BC47 / RB47 | GTGCAACTTTCCCACAGGTAGTTC |
| BC48 / RB48 | CATCTGGAACGTGGTACACCTGTA |
| BC49 / RB49 | ACTGGTGCAGCTTTGAACATCTAG |
| BC50 / RB50 | ATGGACTTTGGTAACTTCCTGCGT |
| BC51 / RB51 | GTTGAATGAGCCTACTGGGTCCTC |
| BC52 / RB52 | TGAGAGACAAGATTGTTCGTGGAC |
| BC53 / RB53 | AGATTCAGACCGTCTCATGCAAAG |
| BC54 / RB54 | CAAGAGCTTTGACTAAGGAGCATG |
| BC55 / RB55 | TGGAAGATGAGACCCTGATCTACG |
| BC56 / RB56 | TCACTACTCAACAGGTGGCATGAA |
| BC57 / RB57 | GCTAGGTCAATCTCCTTCGGAAGT |
| BC58 / RB58 | CAGGTTACTCCTCCGTGAGTCTGA |
| BC59 / RB59 | TCAATCAAGAAGGGAAAGCAAGGT |
| BC60 / RB60 | CATGTTCAACCAAGGCTTCTATGG |
| BC61 / RB61 | AGAGGGTACTATGTGCCTCAGCAC |
| BC62 / RB62 | CACCCACACTTACTTCAGGACGTA |
| BC63 / RB63 | TTCTGAAGTTCCTGGGTCTTGAAC |
| BC64 / RB64 | GACAGACACCGTTCATCGACTTTC |
| BC65 / RB65 | TTCTCAGTCTTCCTCCAGACAAGG |
| BC66 / RB66 | CCGATCCTTGTGGCTTCTAACTTC |
| BC67 / RB67 | GTTTGTCATACTCGTGTGCTCACC |
| BC68 / RB68 | GAATCTAAGCAAACACGAAGGTGG |
| BC69 / RB69 | TACAGTCCGAGCCTCATGTGATCT |
| BC70 / RB70 | ACCGAGATCCTACGAATGGAGTGT |
| BC71 / RB71 | CCTGGGAGCATCAGGTAGTAACAG |
| BC72 / RB72 | TAGCTGACTGTCTTCCATACCGAC |
| BC73 / RB73 | AAGAAACAGGATGACAGAACCCTC |
| BC74 / RB74 | TACAAGCATCCCAACACTTCCACT |
| BC75 / RB75 | GACCATTGTGATGAACCCTGTTGT |
| BC76 / RB76 | ATGCTTGTTACATCAACCCTGGAC |
| BC77 / RB77 | CGACCTGTTTCTCAGGGATACAAC |
| BC78 / RB78 | AACAACCGAACCTTTGAATCAGAA |
| BC79 / RB79 | TCTCGGAGATAGTTCTCACTGCTG |
| BC80 / RB80 | CGGATGAACATAGGATAGCGATTC |
| BC81 / RB81 | CCTCATCTTGTGAAGTTGTTTCGG |
| BC82 / RB82 | ACGGTATGTCGAGTTCCAGGACTA |
| BC83 / RB83 | TGGCTTGATCTAGGTAAGGTCGAA |
| BC84 / RB84 | GTAGTGGACCTAGAACCTGTGCCA |
| BC85 / RB85 | AACGGAGGAGTTAGTTGGATGATC |
| BC86 / RB86 | AGGTGATCCCAACAAGCGTAAGTA |
| BC87 / RB87 | TACATGCTCCTGTTGTTAGGGAGG |
| BC88 / RB88 | TCTTCTACTACCGATCCGAAGCAG |
| BC89 / RB89 | ACAGCATCAATGTTTGGCTAGTTG |
| BC90 / RB90 | GATGTAGAGGGTACGGTTTGAGGC |
| BC91 / RB91 | GGCTCCATAGGAACTCACGCTACT |
| BC92 / RB92 | TTGTGAGTGGAAAGATACAGGACC |
| BC93 / RB93 | AGTTTCCATCACTTCAGACTTGGG |
| BC94 / RB94 | GATTGTCCTCAAACTGCCACCTAC |
| BC95 / RB95 | CCTGTCTGGAAGAAGAATGGACTT |
| BC96 / RB96 | CTGAACGGTCATAGAGTCCACCAT |
3. End-prep
材料
- 100 ng of each sheared DNA sample to be barcoded in 45 µl
耗材
- NEBNext® Ultra II 末端修复模块(NEB, E7546)中提供的 NEBNext Ultra II末端修复酶混合物(End Prep Enzyme Mix )
- NEBNext® Ultra II 末端修复模块(NEB, E7546)中提供的 NEBNext Ultra II末端修复反应缓冲液(Prep Reaction Buffer)
- 新制备的 80% 乙醇(用无核酸酶水配制)
- 无核酸酶水(如ThermoFisher,AM9937)
- Agencourt AMPure XP beads (Beckman Coulter, A63881)
- 0.2 ml 薄壁 PCR 离心管 或 0.2 ml 96 孔 PCR 板
- Qubit™ 分析管(Invitrogen, Q32856)
- Qubit dsDNA HS Assay(双链DNA高灵敏度检测)试剂盒(Invitrogen, Q32851)
仪器
- P1000 移液枪和枪头
- P100 移液枪和枪头
- P10 移液枪和枪头
- 热循环仪
- 盛有冰的冰桶
- 迷你离心机
- 磁力架
- 涡旋混匀仪
可选仪器
- Qubit™ 荧光计(或用于质控检测的等效仪器)
Check your flow cell.
We recommend performing a flow cell check before starting your library prep to ensure you have a flow cell with enough pores for a good sequencing run.
See the flow cell check document for more information.
Prepare the NEBNext Ultra II End Repair / dA-tailing Module reagents in accordance with manufacturer's instructions, and place on ice:
For optimal performance, NEB recommend the following:
Thaw all reagents on ice.
Ensure the reagents are well mixed.
Note: Do not vortex the Ultra II End Prep Enzyme Mix.Always spin down tubes before opening for the first time each day.
The NEBNext Ultra II End Prep Reaction Buffer may contain a white precipitate. If this occurs, allow the mixture to come to room temperature and pipette the buffer several times to break up the precipitate, followed by a quick vortex to mix.
Prepare the DNA in nuclease-free water.
- Transfer 100 ng DNA of each sample into a fresh 0.2 ml PCR tube or plate.
- Adjust the volume to 45 μl with nuclease-free water.
- Mix thoroughly by flicking the tube to avoid unwanted shearing.
- Spin down briefly in a microfuge.
Set up the end-repair reaction as follows for each library:
| Reagent | Volume per sample |
|---|---|
| 100 ng DNA | 45 µl |
| Ultra II End-prep reaction buffer | 7 µl |
| Ultra II End-prep enzyme mix | 3 µl |
| Nuclease-free water | 5 µl |
| Total | 60 µl |
Mix by pipetting and briefly spin down.
Using a thermal cycler, incubate for 5 minutes at 20 °C and 5 minutes at 65 °C. Then cool down to between 4°C and 20°C on the thermal cycler or place the samples on ice.
Resuspend the AMPure XP beads by vortexing.
Add 60 µl of resuspended AMPure XP beads to the end-prep reaction and mix by pipetting.
Incubate at room temperature for 5 minutes.
Freshly prepare sufficient 80% ethanol in nuclease-free water for all of your samples. Allow enough for 400 µl per sample, with some excess.
Spin down the samples and pellet on a magnet until supernatant is clear and colourless. Keep the samples on the magnet, and pipette off the supernatant.
Keep the samples on the magnet and wash the beads with 200 µl of freshly prepared 80% ethanol without disturbing the pellet. Remove the ethanol using a pipette and discard.
Repeat the previous step.
Spin down and place the samples back on the magnetic rack. Pipette off any residual ethanol. Allow the pellet to dry for ~30 seconds, but do not dry the pellet to the point of cracking.
Remove the samples from the magnet and resuspend each pellet in 16 µl nuclease-free water by gently pipetting up and down or by flicking the tube. Incubate for 2 minutes at room temperature.
Pellet the beads on a magnet until the eluate is clear and colourless.
Remove eluate once it is clear and colourless. Transfer each eluted sample to a new tube or plate well.
Quantify 1 µl of end-prepped DNA using a Qubit fluorometer.
Take forward the end-prepped DNA into the next step. However, at this point it is also possible to store the sample at 4°C overnight.
4. Ligation of Barcode Adapter
材料
- Barcode Adapter (BCA)
耗材
- NEB Blunt/TA Ligase Master Mix (NEB, cat # M0367)
- Agencourt AMPure XP beads (Beckman Coulter, A63881)
- 无核酸酶水(如ThermoFisher,AM9937)
- 新制备的 80% 乙醇(用无核酸酶水配制)
- 0.2 ml 薄壁 PCR 离心管 或 0.2 ml 96 孔 PCR 板
- Qubit™ 分析管(Invitrogen, Q32856)
- Qubit dsDNA HS Assay(双链DNA高灵敏度检测)试剂盒(Invitrogen, Q32851)
仪器
- 迷你离心机
- Hula混匀仪(低速旋转式混匀仪)
- 涡旋混匀仪
- 盛有冰的冰桶
- 多通道移液枪和枪头
- 磁力架
- P1000 移液枪和枪头
- P100 移液枪和枪头
- P10 移液枪和枪头
- Qubit™ 荧光计(或用于质控检测的等效仪器)
Prepare the NEB Blunt/TA Ligase Master Mix according to the manufacturer's instructions, and place on ice:
Thaw the reagents at room temperature.
Spin down the reagent tubes for 5 seconds.
Ensure the reagents are fully mixed by performing 10 full volume pipette mixes.
Spin down the Barcode Adapter (BCA), pipette mix and place on ice.
Add the reagents in the order given below, into fresh 0.2 ml PCR tubes or 96-well plate:
| Reagent | Volume |
|---|---|
| End-prepped DNA | 15 µl |
| Barcode Adapter | 10 µl |
| Blunt/TA Ligase Master Mix | 25 µl |
| Total | 50 µl |
Mix by pipetting and briefly spin down.
Incubate the samples for 10 minutes at room temperature.
Resuspend the AMPure XP beads by vortexing.
Add 20 µl of resuspended AMPure XP beads to each sample for a 0.4X clean and mix by pipetting up and down ten times.
Incubate on a Hula mixer (rotator mixer) for 5 minutes at room temperature.
Freshly prepare sufficient 80% ethanol in nuclease-free water for all of your samples. Allow enough for 400 µl per sample, with some excess.
Place on a magnetic rack, allow beads to pellet and pipette off supernatant.
Keep the samples on the magnet and wash the beads with 200 µl of freshly prepared 80% ethanol without disturbing the pellet. Remove the ethanol using a pipette and discard.
Repeat the previous step.
Place the samples back on the magnet. Pipette off any residual 80% ethanol. Allow to dry for ~30 seconds, but do not dry the pellet to the point of cracking.
Remove the samples from the magnet and resuspend pellet in 25 µl nuclease-free water. Incubate for 2 minutes at room temperature.
Pellet the beads on a magnet until the eluate is clear and colourless.
Remove and retain the eluate once it is clear and colourless. Transfer each eluted sample to a fresh 0.2 ml PCR tube or plate.
- Dispose of the pelleted beads.
Quantify 1 µl of the adapter ligated DNA using a Qubit fluorometer.
Take forward the adapter ligated samples into the Barcoding PCR step. However, at this point it is also possible to store the sample at 4°C overnight.
5. Barcoding PCR
材料
- 100-200 fmol of each adapter-ligated sample
- OR 100-200 fmol of each amplicon sample
- PCR Barcodes (BC01-96, at 10 µM)
耗材
- LongAmp 热启动 Taq 酶 2X 预混液(NEB, M0533)
- 无核酸酶水(如ThermoFisher,AM9937)
- Agencourt AMPure XP 磁珠(Beckman Coulter™, A63881)
- 新制备的 80% 乙醇(用无核酸酶水配制)
- 1.5 ml Eppendorf DNA LoBind 离心管
- 0.2 ml 薄壁 PCR 离心管 或 0.2 ml 96 孔 PCR 板
- Qubit™ 分析管(Invitrogen, Q32856)
- Qubit dsDNA HS Assay(双链DNA高灵敏度检测)试剂盒(Invitrogen, Q32851)
仪器
- 热循环仪
- 磁力架
- 迷你离心机
- P1000 移液枪和枪头
- P100 移液枪和枪头
- P10 移液枪和枪头
- Qubit™ 荧光计(或用于质控检测的等效仪器)
Please note, this protocol is written for a template input of 100–200 fmol with PCR Barcodes (BC01-96) used at a final concentration of 0.2 µM. However, the input mass and the number of PCR cycles may be adjusted as appropriate depending on the requirements of the experiment.
Thaw the PCR Barcodes (BC01-96) required for your number of samples at room temperature. Individually mix the barcodes by pipetting, spin down, and place on ice.
If using amplicon samples, ensure the samples have undergone a round of PCR with tailed primers before commencing with the protocol.
Prepare the samples in nuclease-free water:
- Transfer 100-200 fmol of each sample to a clear 0.2 ml PCR tube or plate.
- For 1–12 samples: Adjust the volume to 48 μl with nuclease-free water
- For 13–96 samples: Adjust the volume to 24 μl with nuclease-free water
- Mix thoroughly by flicking the tube or plate to avoid unwanted shearing.
- Spin down briefly in a microfuge.
Select a unique barcode for each sample to be run together on the same flow cell.
Note: Only use one barcode per sample.
Set up a barcoding PCR reaction as follows for each library in fresh 0.2 ml PCR tubes or plate:
Between each addition, pipette mix 10-20 times.
| Reagent | Volume per sample for using 1–12 barcodes | Volume per sample for using 13 barcodes or more |
|---|---|---|
| PCR Barcode (one of BC1-BC96, at 10 µM) | 2 µl | 1 µl |
| Adapter ligated DNA or amplicons with tailed primers | 48 µl | 24 µl |
| HotStart LongAmp Taq 2x master mix | 50 µl | 25 µl |
| Total volume | 100 µl | 50 µl |
If the amount of input material is altered, the number of PCR cycles may need to be adjusted to produce the same yield.
Mix by pipetting and briefly spin down.
Amplify using the following cycling conditions:
| Cycle step | Temperature | Time | No. of cycles |
|---|---|---|---|
| Initial denaturation | 94 °C | 3 mins | 1 |
| Denaturation | 94 °C | 15 secs | 15-18 (a) |
| Annealing | 56 °C | 15 secs | 15-18 (a) |
| Extension | 65 °C | 6 mins (b) | 15-18 (a) |
| Final extension | 65 °C | 10 mins | 1 |
| Hold | 4 °C | ∞ |
a. Adjust accordingly if input quantities are altered.
b. Adjust accordingly for different lengths of amplicons and the type of polymerase that is being used (LongAmp Taq amplifies at a rate of 50 seconds per kb). Here 6 min is used for ~8 kb templates.
Resuspend the AMPure XP beads by vortexing.
Add 0.4X volume of resuspended AMPure XP Beads to each reaction and mix by flicking the tube.
| Reagent | Volume for 100 µl samples | Volume for 50 µl samples |
|---|---|---|
| AMPure XP Beads | 40 µl | 20 µl |
Incubate at room temperature for 5 minutes.
Freshly prepare sufficient 80% ethanol in nuclease-free water for all of your samples. Allow enough for 400 µl per sample, with some excess.
Place on a magnetic rack, allow beads to pellet and pipette off supernatant.
Keep the samples on the magnet and wash the beads with 200 µl of freshly prepared 80% ethanol without disturbing the pellets. Remove the ethanol using a pipette and discard.
Repeat the previous step.
Spin down and place the samples back on the magnet. Pipette off any residual ethanol. Allow to dry for ~30 seconds, but do not dry the pellets to the point of cracking.
Remove the samples from the magnetic rack and resuspend each pellet in 25 µl nuclease-free water. Incubate for 2 minutes at room temperature.
Pellet the beads on a magnetic rack until the eluate is clear and colourless.
Remove and retain 25 µl of each eluate into clean 0.2 ml PCR tubes or plate.
Dispose of the pelleted beads
Quantify the barcoded library using a Qubit fluorometer and pool all barcoded libraries in the desired ratios in a 1.5 ml DNA LoBind Eppendorf tube.
Gel analysis of amplified and ligated DNA
Sometimes a high-molecular weight product is visible in the wells of the gel when the PCR products are run, instead of the expected smear. These libraries are typically associated with poor sequencing performance. We have found that repeating the PCR with fewer cycles can remedy this.
Prepare 1 µg of pooled barcoded libraries in 50 µl nuclease-free water.
If the volume of your pool exceeds the 50 µl required for the end-prep reaction, consider a 2.5X AMPure XP Bead purification of the pool to concentrate your sample.
This pooled library is now ready to be end-repaired and adapted for sequencing. However, at this point it is also possible to store the sample at 4°C overnight.
6. End-prep
材料
- AMPure XP 磁珠(AXP)
耗材
- NEBNext® Ultra II 末端修复模块(NEB, E7546)中提供的 NEBNext Ultra II末端修复酶混合物(End Prep Enzyme Mix )
- NEBNext® Ultra II 末端修复模块(NEB, E7546)中提供的 NEBNext Ultra II末端修复反应缓冲液(Prep Reaction Buffer)
- 0.2 ml 薄壁PCR管
- 1.5 ml Eppendorf DNA LoBind 离心管
- 无核酸酶水(如ThermoFisher,AM9937)
- 新制备的 80% 乙醇(用无核酸酶水配制)
- Qubit™ 分析管(Invitrogen, Q32856)
- Qubit dsDNA HS Assay(双链DNA高灵敏度检测)试剂盒(Invitrogen, Q32851)
仪器
- P1000 移液枪和枪头
- P100 移液枪和枪头
- P10 移液枪和枪头
- 热循环仪
- 迷你离心机
- Hula混匀仪(低速旋转式混匀仪)
- 磁力架
- 盛有冰的冰桶
可选仪器
- Qubit™ 荧光计(或用于质控检测的等效仪器)
Prepare the NEBNext Ultra II End Repair / dA-tailing Module reagents in accordance with manufacturer's instructions, and place on ice:
For optimal performance, NEB recommend the following:
Thaw all reagents on ice.
Ensure the reagents are well mixed.
Note: Do not vortex the Ultra II End Prep Enzyme Mix.Always spin down tubes before opening for the first time each day.
The NEBNext Ultra II End Prep Reaction Buffer may contain a white precipitate. If this occurs, allow the mixture to come to room temperature and pipette the buffer several times to break up the precipitate, followed by a quick vortex to mix.
Prepare the PCR amplified DNA in nuclease-free water
- Transfer 1 ug genomic DNA into a 0.2 ml thin-walled PCR tube.
- Adjust the volume to 50 μl with nuclease-free water.
- Mix thoroughly by flicking the tube to avoid unwanted shearing.
- Spin down briefly in a microfuge.
In a 0.2 ml thin-walled PCR tube, mix the following:
| Reagent | Volume |
|---|---|
| DNA | 50 µl |
| Ultra II End-prep Reaction Buffer | 7 µl |
| Ultra II End-prep Enzyme Mix | 3 µl |
| Total | 60 µl |
Thoroughly mix the reaction by gently pipetting and briefly spinning down.
Using a thermal cycler, incubate at 20°C for 5 minutes and 65°C for 5 minutes. Then cool down to between 4°C and 20°C on the thermal cycler or place the samples on ice.
Resuspend the AMPure XP Beads (AXP) by vortexing.
Transfer the DNA sample to a clean 1.5 ml Eppendorf DNA LoBind tube.
Add 60 µl of resuspended the AMPure XP Beads (AXP) to the end-prep reaction and mix by flicking the tube.
Incubate on a Hula mixer (rotator mixer) for 5 minutes at room temperature.
Freshly prepare 500 μl of 80% ethanol in nuclease-free water.
Spin down the sample and pellet on a magnet until supernatant is clear and colourless. Keep the tube on the magnet, and pipette off the supernatant.
Keep the tube on the magnet and wash the beads with 200 µl of freshly prepared 80% ethanol without disturbing the pellet. Remove the ethanol using a pipette and discard.
Repeat the previous step.
Spin down and place the tube back on the magnet. Pipette off any residual ethanol. Allow to dry for ~30 seconds, but do not dry the pellet to the point of cracking.
Remove the tube from the magnetic rack and resuspend the pellet in 61 µl nuclease-free water by gently pipetting up and down or by flicking the tube. Incubate for 2 minutes at room temperature.
Pellet the beads on a magnet until the eluate is clear and colourless, for at least 1 minute.
Remove and retain 61 µl of eluate into a clean 1.5 ml Eppendorf DNA LoBind tube.
Quantify 1 µl of eluted sample using a Qubit fluorometer.
Take forward the repaired and end-prepped DNA into the adapter ligation step. However, at this point it is also possible to store the sample at 4°C overnight.
7. Adapter ligation and clean-up
材料
- 连接接头(LA)
- 连接测序试剂盒内的连接缓冲液(LNB)
- 长片段缓冲液(LFB)
- 短片段缓冲液(SFB)
- AMPure XP 磁珠(AXP)
- 洗脱缓冲液(EB)
耗材
- 耐盐T4 DNA连接酶(NEB, M0467)
- 1.5 ml Eppendorf DNA LoBind 离心管
- Qubit dsDNA HS Assay(双链DNA高灵敏度检测)试剂盒(Invitrogen, Q32851)
- Qubit™ 分析管(Invitrogen, Q32856)
仪器
- 磁力架
- 迷你离心机
- 涡旋混匀仪
- P1000 移液枪和枪头
- P100 移液枪和枪头
- P20 移液枪和枪头
- P10 移液枪和枪头
- Qubit™ 荧光计(或用于质控检测的等效仪器)
We recommend using the Salt-T4® DNA Ligase (NEB, M0467).
Salt-T4® DNA Ligase (NEB, M0467) can be bought separately or is provided in the NEBNext® Companion Module v2 for Oxford Nanopore Technologies® Ligation Sequencing (NEB, E7672S or E7672L).
The Quick T4 DNA Ligase (NEB, E6057) available in the previous version NEBNext® Companion Module for Oxford Nanopore Technologies® Ligation Sequencing (NEB, E7180S or E7180L) is also compatible, but the new recommended reagent offers more efficient end ligation.
Although third-party ligase products may be supplied with their own buffer, the ligation efficiency of the Ligation Adapter (LA) is higher when using the Ligation Buffer (LNB) supplied in the Ligation Sequencing Kit.
Spin down the Ligation Adapter (LA) and Salt-T4® DNA Ligase, and place on ice.
Thaw Ligation Buffer (LNB) at room temperature, spin down and mix by pipetting. Due to viscosity, vortexing this buffer is ineffective. Place on ice immediately after thawing and mixing.
Thaw the Elution Buffer (EB) at room temperature and mix by vortexing. Then spin down and place on ice.
Depending on the wash buffer (LFB or SFB) used, the clean-up step after adapter ligation is designed to either enrich for DNA fragments of >3 kb, or purify all fragments equally.
- To enrich for DNA fragments of 3 kb or longer, use Long Fragment Buffer (LFB).
- To retain DNA fragments of all sizes, use Short Fragment Buffer (SFB).
Thaw either Long Fragment Buffer (LFB) or Short Fragment Buffer (SFB) at room temperature and mix by vortexing. Then spin down and keep at room temperature.
In a 1.5 ml Eppendorf DNA LoBind tube, mix in the following order:
Between each addition, pipette mix 10-20 times.
| Reagent | Volume |
|---|---|
| DNA sample from the previous step | 60 µl |
| Ligation Adapter (LA) | 5 µl |
| Ligation Buffer (LNB) | 25 µl |
| Salt-T4® DNA Ligase | 10 µl |
| Total | 100 µl |
Thoroughly mix the reaction by gently pipetting and briefly spinning down.
Incubate the reaction for 10 minutes at room temperature.
Resuspend the AMPure XP Beads (AXP) by vortexing.
Add 40 µl of resuspended AMPure XP Beads (AXP) to the reaction and mix by flicking the tube.
Incubate on a Hula mixer (rotator mixer) for 5 minutes at room temperature.
Spin down the sample and pellet on a magnet. Keep the tube on the magnet, and pipette off the supernatant when clear and colourless.
Wash the beads by adding either 250 μl Long Fragment Buffer (LFB) or 250 μl Short Fragment Buffer (SFB). Flick the beads to resuspend, spin down, then return the tube to the magnetic rack and allow the beads to pellet. Remove the supernatant using a pipette and discard.
Repeat the previous step.
Spin down and place the tube back on the magnet. Pipette off any residual supernatant. Allow to dry for ~30 seconds, but do not dry the pellet to the point of cracking.
Remove the tube from the magnetic rack and resuspend the pellet in 15 µl Elution Buffer (EB). Spin down and incubate for 10 minutes at room temperature. For high molecular weight DNA, incubating at 37°C can improve the recovery of long fragments.
Pellet the beads on a magnet until the eluate is clear and colourless, for at least 1 minute.
Remove and retain 15 µl of eluate containing the DNA library into a clean 1.5 ml Eppendorf DNA LoBind tube.
Dispose of the pelleted beads
Quantify 1 µl of eluted sample using a Qubit fluorometer.
Depending on your DNA library fragment size, prepare your final library in 12 µl of Elution Buffer (EB).
| Fragment library length | Flow cell loading amount |
|---|---|
| Very short (<1 kb) | 100 fmol |
| Short (1-10 kb) | 35–50 fmol |
| Long (>10 kb) | 300 ng |
Note: If the library yields are below the input recommendations, load the entire library.
If required, we recommend using a mass to mol calculator such as the NEB calculator.
The prepared library is used for loading into the flow cell. Store the library on ice or at 4°C until ready to load.
Library storage recommendations
We recommend storing libraries in Eppendorf DNA LoBind tubes at 4°C for short-term storage or repeated use, for example, re-loading flow cells between washes. For single use and long-term storage of more than 3 months, we recommend storing libraries at -80°C in Eppendorf DNA LoBind tubes.
8. Priming and loading the MinION and GridION Flow Cell
材料
- 测序芯片冲洗液(FCF)
- 测序芯片系绳(FCT)
- 文库溶液(LIS)
- 文库颗粒(LIB)
- 测序缓冲液(SB)
耗材
- MinION/GridION测序芯片
- 1.5 ml Eppendorf DNA LoBind 离心管
- 无核酸酶水(如ThermoFisher,AM9937)
- 牛血清白蛋白(BSA)(50 mg/mL)(例如 Invitrogen™ UltraPure™ BSA 50 mg/mL, AM2616)
仪器
- MinION 或 GridION 测序仪
- MinION 及GridION 测序芯片遮光片
- P1000 移液枪和枪头
- P100 移液枪和枪头
- P20 移液枪和枪头
- P10 移液枪和枪头
Please note, this kit is only compatible with R10.4.1 flow cells (FLO-MIN114).
从冰箱内取出测序芯片,室温放置 20 分钟,可在预处理和上样时更清晰地看到传感器阵列。
Priming and loading a flow cell
We recommend that you watch the 'Priming and loading your flow cell' video before your first run.
Thaw the Sequencing Buffer (SB), Library Beads (LIB) or Library Solution (LIS, if using), Flow Cell Tether (FCT) and Flow Cell Flush (FCF) at room temperature before mixing by vortexing. Then spin down and store on ice.
For optimal sequencing performance and improved output on MinION R10.4.1 flow cells (FLO-MIN114), add Bovine Serum Albumin (BSA) to the flow cell priming mix at a final concentration of 0.2 mg/ml.
Note: We do not recommend using any other albumin type (e.g. recombinant human serum albumin).
To prepare the flow cell priming mix with BSA, combine Flow Cell Flush (FCF) and Flow Cell Tether (FCT), as directed below. Mix by pipetting at room temperature.
In a suitable tube for the number of flow cells, combine the following reagents:
| Reagent | Volume per flow cell |
|---|---|
| Flow Cell Flush (FCF) | 1,170 µl |
| Bovine Serum Albumin (BSA) at 50 mg/ml | 5 µl |
| Flow Cell Tether (FCT) | 30 µl |
| Total volume | 1,205 µl |
Open the MinION or GridION device lid and slide the flow cell under the clip. Press down firmly on the priming port cover to ensure correct thermal and electrical contact.
Complete a flow cell check to assess the number of pores available before loading the library.
This step can be omitted if the flow cell has been checked previously.
See the flow cell check document for more information.
Slide the flow cell priming port cover clockwise to open the priming port.
Take care when drawing back buffer from the flow cell. Do not remove more than 20-30 µl, and make sure that the array of pores are covered by buffer at all times. Introducing air bubbles into the array can irreversibly damage pores.
After opening the priming port, check for a small air bubble under the cover. Draw back a small volume to remove any bubbles:
- Set a P1000 pipette to 200 µl
- Insert the tip into the priming port
- Turn the wheel until the dial shows 220-230 µl, to draw back 20-30 µl, or until you can see a small volume of buffer entering the pipette tip
Note: Visually check that there is continuous buffer from the priming port across the sensor array.
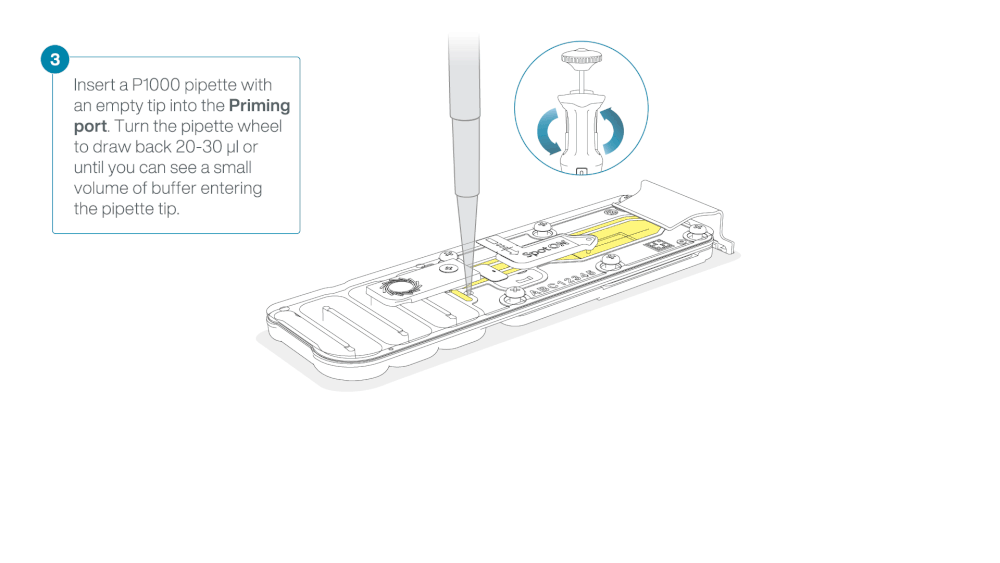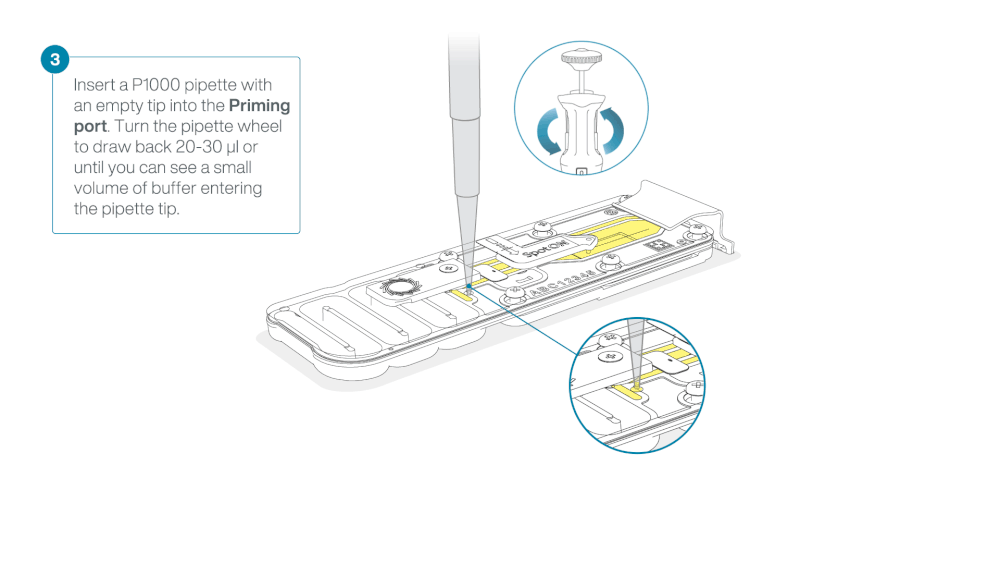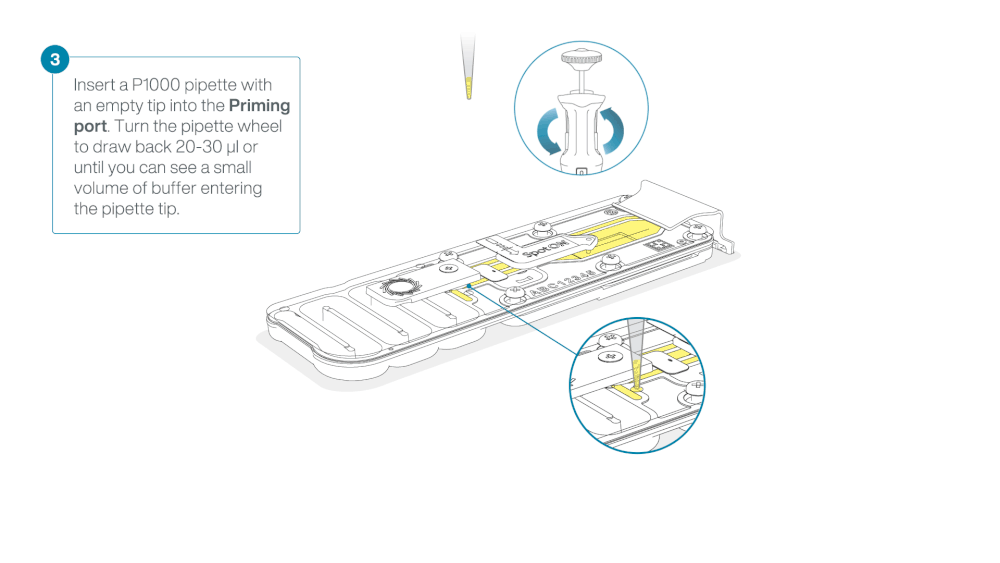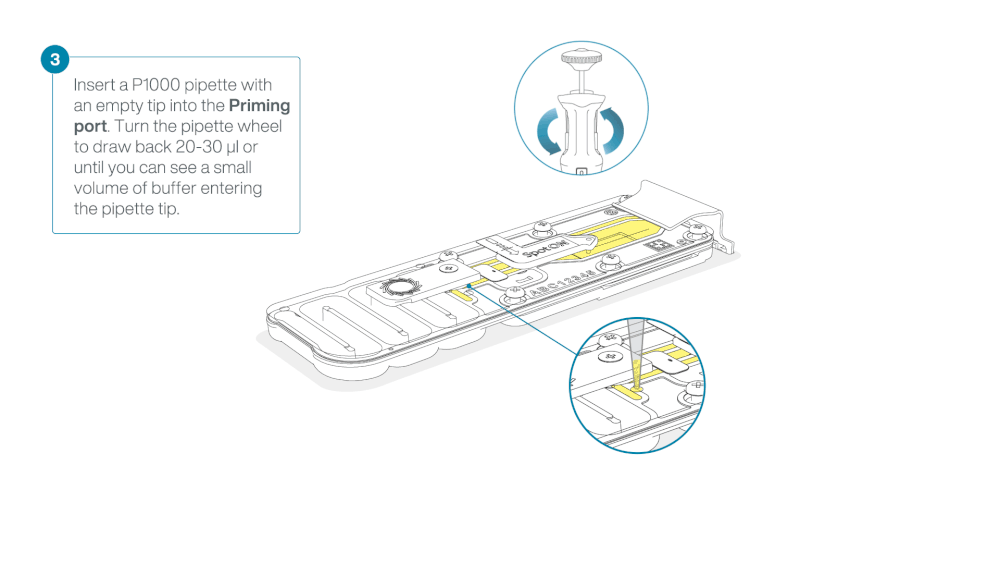
Load 800 µl of the priming mix into the flow cell via the priming port, avoiding the introduction of air bubbles. Wait for five minutes. During this time, prepare the library for loading by following the steps below.

Thoroughly mix the contents of the Library Beads (LIB) by pipetting.
The Library Beads (LIB) tube contains a suspension of beads. These beads settle very quickly. It is vital that they are mixed immediately before use.
We recommend using the Library Beads (LIB) for most sequencing experiments. However, the Library Solution (LIS) is available for more viscous libraries.
In a new 1.5 ml Eppendorf DNA LoBind tube, prepare the library for loading as follows:
| Reagent | Volume per flow cell |
|---|---|
| Sequencing Buffer (SB) | 37.5 µl |
| Library Beads (LIB) mixed immediately before use, or Library Solution (LIS), if using | 25.5 µl |
| DNA library | 12 µl |
| Total | 75 µl |
Complete the flow cell priming:
- Gently lift the SpotON sample port cover to make the SpotON sample port accessible.
- Load 200 µl of the priming mix into the flow cell priming port (not the SpotON sample port), avoiding the introduction of air bubbles.

Mix the prepared library gently by pipetting up and down just prior to loading.
Add 75 μl of the prepared library to the flow cell via the SpotON sample port in a dropwise fashion. Ensure each drop flows into the port before adding the next.

Gently replace the SpotON sample port cover, making sure the bung enters the SpotON port and close the priming port.

For optimal sequencing output, install the light shield on your flow cell as soon as the library has been loaded.
We recommend leaving the light shield on the flow cell when library is loaded, including during any washing and reloading steps. The shield can be removed when the library has been removed from the flow cell.
Place the light shield onto the flow cell, as follows:
Carefully place the leading edge of the light shield against the clip. Note: Do not force the light shield underneath the clip.
Gently lower the light shield onto the flow cell. The light shield should sit around the SpotON cover, covering the entire top section of the flow cell.

The MinION Flow Cell Light Shield is not secured to the flow cell. Therefore, careful handling is required after installation.
Close the device lid and set up a sequencing run on MinKNOW.
When a flow cell is inserted into the MinION Mk1D, the device lid will sit on top of the flow cell, leaving a small gap around the sides. This is normal and has no impact on the performance of the device.
Please refer to this FAQ regarding the device lid.

9. Data acquisition and basecalling
How to start sequencing
Once you have loaded your flow cell, the sequencing run can be started on MinKNOW, our sequencing software that controls the device, data acquisition and real-time basecalling. For more detailed information on setting up and using MinKNOW, please see the MinKNOW protocol.
MinKNOW can be used and set up to sequence in multiple ways:
- On a computer either directly or remotely connected to a sequencing device.
- Directly on a GridION or PromethION 24/48 sequencing device.
For more information on using MinKNOW on a sequencing device, please see the device user manuals:
To start a sequencing run on MinKNOW:
1. Navigate to the start page and click Start sequencing.
2. Fill in your experiment details, such as name and flow cell position and sample ID.
3. Select the Ligation Sequencing Kit V14 (SQK-LSK114) for the kit and the PCR Barcoding Expansion (EXP-PBC001 or EXP-PBC096) for the Expansion pack on the Kit selection page.
4. Configure the sequencing and output parameters for your sequencing run or keep to the default settings on the Run configuration tab.
Note: If basecalling was turned off when a sequencing run was set up, basecalling can be performed post-run on MinKNOW. For more information, please see the MinKNOW protocol.
5. Click Start to initiate the sequencing run.
Data analysis after sequencing
After sequencing has completed on MinKNOW, the flow cell can be reused or returned, as outlined in the Flow cell reuse and returns section.
After sequencing and basecalling, the data can be analysed. For further information about options for basecalling and post-basecalling analysis, please refer to the Data Analysis document.
In the Downstream analysis section, we outline further options for analysing your data.
10. Flow cell reuse and returns
材料
- 测序芯片清洗剂盒(EXP-WSH004)
After your sequencing experiment is complete, if you would like to reuse the flow cell, please follow the Flow Cell Wash Kit protocol and store the washed flow cell at +2°C to +8°C.
The Flow Cell Wash Kit protocol is available on the Nanopore Community.
We recommend you to wash the flow cell as soon as possible after you stop the run. However, if this is not possible, leave the flow cell on the device and wash it the next day.
Alternatively, follow the returns procedure to send the flow cell back to Oxford Nanopore.
Instructions for returning flow cells can be found here.
If you encounter issues or have questions about your sequencing experiment, please refer to the Troubleshooting Guide that can be found in this protocol.
11. Downstream analysis
Post-basecalling analysis
There are several options for further analysing your basecalled data:
EPI2ME workflows
For in-depth data analysis, Oxford Nanopore Technologies offers a range of bioinformatics tutorials and workflows available in EPI2ME, which are available in the EPI2ME section of the Community. The platform provides a vehicle where workflows deposited in GitHub by our Research and Applications teams can be showcased with descriptive texts, functional bioinformatics code and example data.
Research analysis tools
Oxford Nanopore Technologies' Research division has created a number of analysis tools, that are available in the Oxford Nanopore GitHub repository. The tools are aimed at advanced users, and contain instructions for how to install and run the software. They are provided as-is, with minimal support.
Community-developed analysis tools
If a data analysis method for your research question is not provided in any of the resources above, please refer to the resource centre and search for bioinformatics tools for your application. Numerous members of the Nanopore Community have developed their own tools and pipelines for analysing nanopore sequencing data, most of which are available on GitHub. Please be aware that these tools are not supported by Oxford Nanopore Technologies, and are not guaranteed to be compatible with the latest chemistry/software configuration.
12. Issues during DNA/RNA extraction and library preparation
Below is a list of the most commonly encountered issues, with some suggested causes and solutions.
We also have an FAQ section available on the Nanopore Community Support section.
If you have tried our suggested solutions and the issue still persists, please contact Technical Support via email (support@nanoporetech.com) or via LiveChat in the Nanopore Community.
Low sample quality
| Observation | Possible cause | Comments and actions |
|---|---|---|
| Low DNA purity (Nanodrop reading for DNA OD 260/280 is <1.8 and OD 260/230 is <2.0–2.2) | The DNA extraction method does not provide the required purity | The effects of contaminants are shown in the Contaminants document. Please try an alternative extraction method that does not result in contaminant carryover. Consider performing an additional SPRI clean-up step. |
| Low RNA integrity (RNA integrity number <9.5 RIN, or the rRNA band is shown as a smear on the gel) | The RNA degraded during extraction | Try a different RNA extraction method. For more info on RIN, please see the RNA Integrity Number document. Further information can be found in the DNA/RNA Handling page. |
| RNA has a shorter than expected fragment length | The RNA degraded during extraction | Try a different RNA extraction method. For more info on RIN, please see the RNA Integrity Number document. Further information can be found in the DNA/RNA Handling page. We recommend working in an RNase-free environment, and to keep your lab equipment RNase-free when working with RNA. |
Low DNA recovery after AMPure bead clean-up
| Observation | Possible cause | Comments and actions |
|---|---|---|
| Low recovery | DNA loss due to a lower than intended AMPure beads-to-sample ratio | 1. AMPure beads settle quickly, so ensure they are well resuspended before adding them to the sample. 2. When the AMPure beads-to-sample ratio is lower than 0.4:1, DNA fragments of any size will be lost during the clean-up. |
| Low recovery | DNA fragments are shorter than expected | The lower the AMPure beads-to-sample ratio, the more stringent the selection against short fragments. Please always determine the input DNA length on an agarose gel (or other gel electrophoresis methods) and then calculate the appropriate amount of AMPure beads to use. 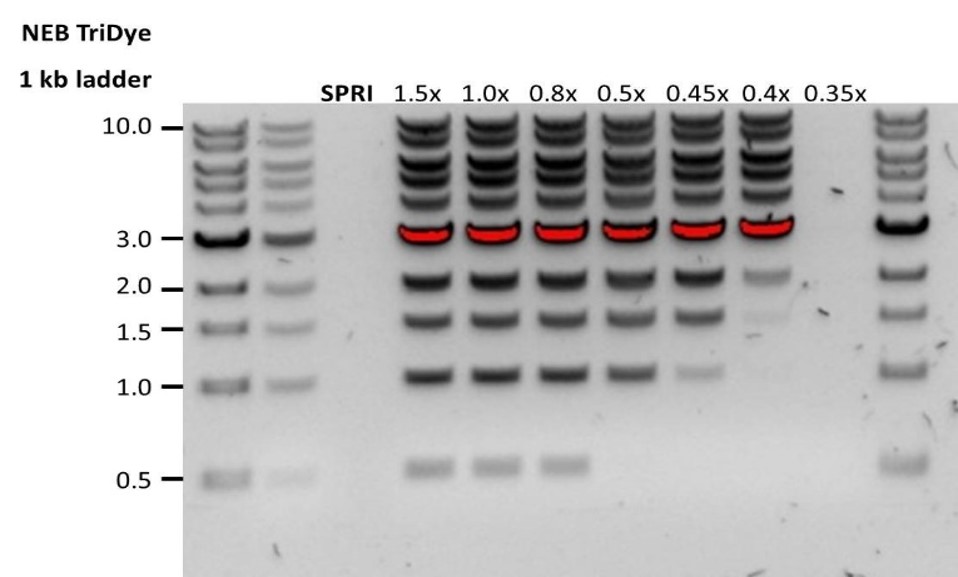 |
| Low recovery after end-prep | The wash step used ethanol <70% | DNA will be eluted from the beads when using ethanol <70%. Make sure to use the correct percentage. |
13. Issues during the sequencing run
Below is a list of the most commonly encountered issues, with some suggested causes and solutions.
We also have an FAQ section available on the Nanopore Community Support section.
If you have tried our suggested solutions and the issue still persists, please contact Technical Support via email (support@nanoporetech.com) or via LiveChat in the Nanopore Community.
Fewer pores at the start of sequencing than after Flow Cell Check
| Observation | Possible cause | Comments and actions |
|---|---|---|
| MinKNOW reported a lower number of pores at the start of sequencing than the number reported by the Flow Cell Check | An air bubble was introduced into the nanopore array | After the Flow Cell Check it is essential to remove any air bubbles near the priming port before priming the flow cell. If not removed, the air bubble can travel to the nanopore array and irreversibly damage the nanopores that have been exposed to air. The best practice to prevent this from happening is demonstrated in videos for how to load a MinION Flow Cell and how to load a PromethION Flow Cell. |
| MinKNOW reported a lower number of pores at the start of sequencing than the number reported by the Flow Cell Check | The flow cell is not correctly inserted into the device | Stop the sequencing run, remove the flow cell from the sequencing device and insert it again, checking that the flow cell is firmly seated in the device and that it has reached the target temperature. If applicable, try a different position on the device (GridION/PromethION). |
| MinKNOW reported a lower number of pores at the start of sequencing than the number reported by the Flow Cell Check | Contaminations in the library damaged or blocked the pores | The pore count during the Flow Cell Check is performed using the QC DNA molecules present in the flow cell storage buffer. At the start of sequencing, the library itself is used to estimate the number of active pores. Because of this, variability of about 10% in the number of pores is expected. A significantly lower pore count reported at the start of sequencing can be due to contaminants in the library that have damaged the membranes or blocked the pores. Alternative DNA/RNA extraction or purification methods may be needed to improve the purity of the input material. The effects of contaminants are shown in the Contaminants Know-how piece. Please try an alternative extraction method that does not result in contaminant carryover. |
MinKNOW script failed
| Observation | Possible cause | Comments and actions |
|---|---|---|
| MinKNOW shows "Script failed" | Restart the computer and then restart MinKNOW. If the issue persists, please collect the MinKNOW log files and contact Technical Support. If you do not have another sequencing device available, we recommend storing the flow cell and the loaded library at 4°C and contact Technical Support for further storage guidance. |
Pore occupancy below 40%
| Observation | Possible cause | Comments and actions |
|---|---|---|
| Pore occupancy <40% | Not enough library was loaded on the flow cell | Ensure the correct volume and concentration as stated on the appropriate protocol for your sequencing library is loaded onto the flow cell. Please quantify the library before loading and calculate fmols using tools like the Promega Biomath Calculator, choosing "dsDNA: µg to fmol" |
| Pore occupancy close to 0 | The Ligation Sequencing Kit was used, and sequencing adapters did not ligate to the DNA | Make sure to use the NEBNext Quick Ligation Module (E6056) and Oxford Nanopore Technologies Ligation Buffer (LNB, provided in the sequencing kit) at the sequencing adapter ligation step, and use the correct amount of each reagent. A Lambda control library can be prepared to test the integrity of the third-party reagents. |
| Pore occupancy close to 0 | The Ligation Sequencing Kit was used, and ethanol was used instead of LFB or SFB at the wash step after sequencing adapter ligation | Ethanol can denature the motor protein on the sequencing adapters. Make sure the LFB or SFB buffer was used after ligation of sequencing adapters. |
| Pore occupancy close to 0 | No tether on the flow cell | Tethers are adding during flow cell priming (FLT tube for Kit 9, 10, 11, FCT for Kit 14, and FTU for ultra-long DNA kits). Make sure FLT/FCT/FTU was added to the buffer (FB for Kit 9, 10, 11, and FCF for Kit 14) before priming. |
Shorter than expected read length
| Observation | Possible cause | Comments and actions |
|---|---|---|
| Shorter than expected read length | Unwanted fragmentation of DNA sample | Read length reflects input DNA fragment length. Input DNA can be fragmented during extraction and library prep. 1. Please review the Extraction Methods in the Nanopore Community for best practice for extraction. 2. Visualise the input DNA fragment length distribution on an agarose gel before proceeding to the library prep. 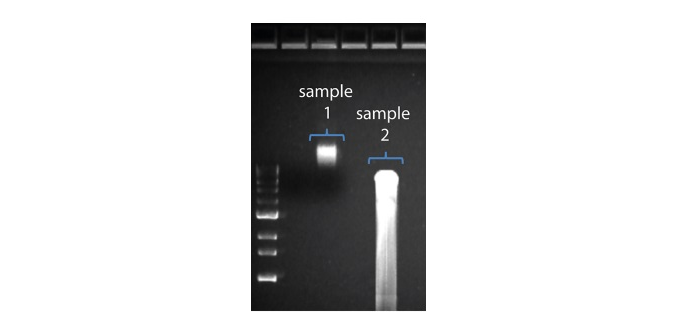 In the image above, Sample 1 is of high molecular weight, whereas Sample 2 has been fragmented. In the image above, Sample 1 is of high molecular weight, whereas Sample 2 has been fragmented.3. During library prep, avoid pipetting and vortexing when mixing reagents. Flicking or inverting the tube is sufficient. |
Large proportion of unavailable pores
| Observation | Possible cause | Comments and actions |
|---|---|---|
Large proportion of unavailable pores (shown as blue in the channels panel and pore activity plot) 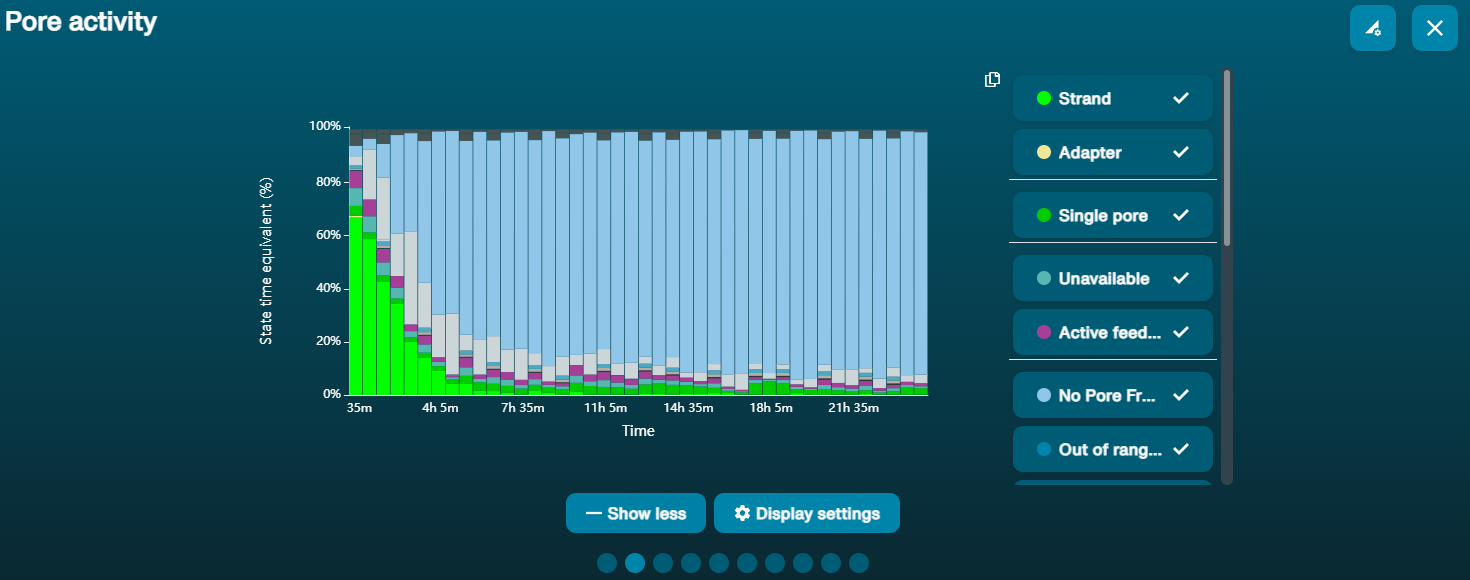 The pore activity plot above shows an increasing proportion of "unavailable" pores over time. The pore activity plot above shows an increasing proportion of "unavailable" pores over time. | Contaminants are present in the sample | Some contaminants can be cleared from the pores by the unblocking function built into MinKNOW. If this is successful, the pore status will change to "sequencing pore". If the portion of unavailable pores stays large or increases: 1. A nuclease flush using the Flow Cell Wash Kit (EXP-WSH004) can be performed, or 2. Run several cycles of PCR to try and dilute any contaminants that may be causing problems. |
Large proportion of inactive pores
| Observation | Possible cause | Comments and actions |
|---|---|---|
| Large proportion of inactive/unavailable pores (shown as light blue in the channels panel and pore activity plot. Pores or membranes are irreversibly damaged) | Air bubbles have been introduced into the flow cell | Air bubbles introduced through flow cell priming and library loading can irreversibly damage the pores. Watch the how to load a MinION Flow Cell or how to load a PromethION Flow Cell videos for best practice. |
| Large proportion of inactive/unavailable pores | Certain compounds co-purified with DNA | Known compounds, include polysaccharides, typically associate with plant genomic DNA. 1. Please refer to the Plant leaf DNA extraction method. 2. Clean-up using the QIAGEN PowerClean Pro kit. 3. Perform a whole genome amplification with the original gDNA sample using the QIAGEN REPLI-g kit. |
| Large proportion of inactive/unavailable pores | Contaminants are present in the sample | The effects of contaminants are shown in the Contaminants Know-how piece. Please try an alternative extraction method that does not result in contaminant carryover. |
Temperature fluctuation
| Observation | Possible cause | Comments and actions |
|---|---|---|
| Temperature fluctuation | The flow cell has lost contact with the device | Check that there is a heat pad covering the metal plate on the back of the flow cell. Re-insert the flow cell and press it down to make sure the connector pins are firmly in contact with the device. If the problem persists, please contact Technical Services. |
Failed to reach target temperature
| Observation | Possible cause | Comments and actions |
|---|---|---|
| MinKNOW shows "Failed to reach target temperature" | The instrument was placed in a location that is colder than normal room temperature, or a location with poor ventilation (which leads to the flow cells overheating) | MinKNOW has a default timeframe for the flow cell to reach the target temperature. Once the timeframe is exceeded, an error message will appear and the sequencing experiment will continue. However, sequencing at an incorrect temperature may lead to a decrease in throughput and lower q-scores. Please adjust the location of the sequencing device to ensure that it is placed at room temperature with good ventilation, then re-start the process in MinKNOW. Please refer to this link for more information on MinION temperature control. |

.gif)







