Solución de secuenciación de aislados microbianos exclusivo de Nanopore (NO-MISS) a partir de cultivos celulares utilizando SQK-RBK114 (.24 o .96) (ISO_9205_v114_revI_14Jan2026)
MinION: Protocol
Solución de secuenciación de aislados microbianos exclusivo de Nanopore (NO-MISS) a partir de cultivos celulares utilizando SQK-RBK114 (.24 o .96) V ISO_9205_v114_revI_14Jan2026
Método completo que describe la extracción de muestras, la preparación de bibliotecas, la secuenciación y el análisis de datos. Este protocolo:
- Utiliza ADN genómico
- Permite multiplexar de 4-24 muestras en MinION o GridION, o 4-96 muestras en PromethION
- Se tarda unos 60 minutos en preparar la biblioteca
- Incluye fragmentación del ADN
- Está optimizado para ofrecer un rendimiento elevado
- Es compatible con las celdas de flujo R10.4.1
De uso exclusivo en investigación
FOR RESEARCH USE ONLY
Contents
Introducción al protocolo
Preparación de muestras
Preparación de la biblioteca
Secuenciación y análisis de datos
- 6. Adquisición de datos e identificación de bases
- 7. Análisis de datos
- 8. Reutilización y devolución de celdas de flujo
Resolución de problemas
Descripción general
Método completo que describe la extracción de muestras, la preparación de bibliotecas, la secuenciación y el análisis de datos. Este protocolo:
- Utiliza ADN genómico
- Permite multiplexar de 4-24 muestras en MinION o GridION, o 4-96 muestras en PromethION
- Se tarda unos 60 minutos en preparar la biblioteca
- Incluye fragmentación del ADN
- Está optimizado para ofrecer un rendimiento elevado
- Es compatible con las celdas de flujo R10.4.1
De uso exclusivo en investigación
1. Descripción general
Introducción al protocolo Solución de secuenciación de aislados microbianos exclusivo de Nanopore (NO-MISS)
Este protocolo completo describe nuestra Solución de secuenciación de aislados microbianos exclusivo de Nanopore (NO-MISS): un enfoque flexible que permite secuenciar de 4 a 24 genomas de aislados microbianos por celda de flujo MinION y generar una cobertura mínima de 50x por genoma.
El umbral de cobertura de 50x es suficiente para realizar los análisis posteriores, entre ellos el ensamblaje y la resolución precisa de plásmidos, la detección de resistencia a los antimicrobianos (AMR), la tipificación multilocus del genoma central (cgMLST) y del genoma completo (wgMLST), así como la tipificación cg/wgSNP. EPI2ME, que proporciona un flujo bioinformático fácil de usar, está disponible para analizar los datos.
Ofrecemos múltiples métodos de extracción de ADN, en función de los requisitos y del organismo de partida (bacterias, hongos/levaduras). Estos son fundamentales para lograr un rendimiento fiable de la celda de flujo y la cobertura del genoma.
Acto seguido, el ADNg se somete a tagmentación y se secuencia con nuestro Rapid Barcoding Kit (SQK-RBK114.24 o SQK-RBK114.96). Es posible procesar hasta 24 muestras por experimento de secuenciación en el caso de aislados bacterianos (genomas de hasta 7 Mb) y hasta 8 muestras en el caso de aislados de hongos/levaduras, con el fin de alcanzar el umbral de cobertura de 50x. Utilice un mínimo de cuatro códigos de barras por ejecución para mantener el rendimiento.
Incluimos también instrucciones detalladas para configurar la secuenciación en MinKNOW y el análisis posterior, con el fin de ofrecer un protocolo completo. Recomendamos secuenciar hasta 72 horas y generar al menos una cobertura de 50x por muestra (aproximadamente 0,5 Gb por código de barras, partiendo de un tamaño genómico de 5 Mb).
Antes de iniciar una un experimento de secuenciación, recomendamos actualizar MinKNOW. El modelo de identificación de bases v4.3, disponible a partir de Dorado 0.5.0, ofrece una mayor precisión en el ADN bacteriano y está incluido en MinKNOW 24.02 o versiones posteriores.
Encontrará más información sobre la actualización de MinKNOW en nuestro protocolo de MinKNOW.
Descripción general del flujo de trabajo completo
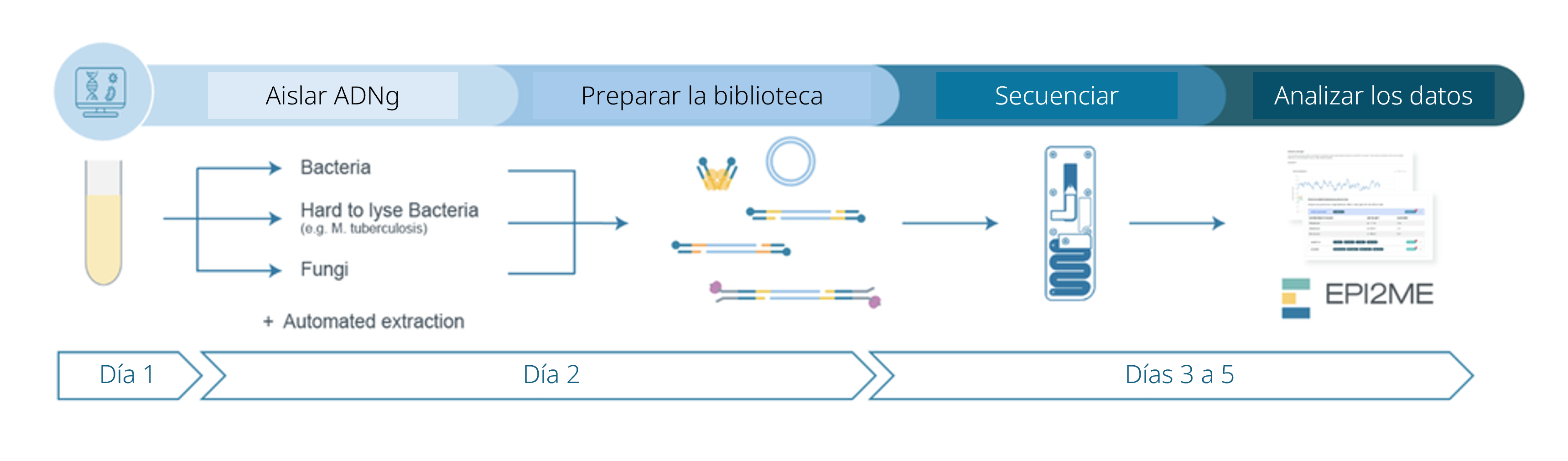
Pasos del flujo de trabajo de secuenciación:
Preparación del experimento
Pasos:
- Extraer el ADN y comprobar su cantidad y la pureza. Los controles de calidad realizados durante el protocolo son esenciales para garantizar el éxito del experimento.
- Disponer del kit de secuenciación, el equipo adecuado y los reactivos de otros fabricantes.
- Descargar el programa informático que obtiene y analiza los datos.
- Comprobar la celda de flujo y asegurarse de que tiene poros suficientes para realizar una buena secuenciación.
Preparación de la muestra
Mediante el método de extracción de ADNg pertinente, deberá lisar las células, extraer el ADNg y cuantificarlo:
- Método general de homogeneización con microesferas:
- Extracción general de ADNg mediante homogeneización con microesferas:
- Indicado en casos de elevado rendimiento y aplicaciones generales.
- Métodos manuales de extracción mediante columnas de sílice:
- Extracción de ADN genómico bacteriano:
- Bacterias como Escherichia coli, Klebsiella pneumoniae, Pseudomonas aeruginosa, Serratia marcescens, Enterococcus faecalis y Bacillus subtilis, o estafilococos como Staphylococcus aureus y Staphylococcus epidermidis.
- Extracción de ADNg de organismos difíciles de lisar:
- Bacterias como Mycobacterium tuberculosis.
- Extracción de ADN genómico de hongos:
- Hongos/levaduras, como Candida albicans, Candida tropicalis y Candida parapsilosis.
Preparación de la biblioteca
La siguiente tabla es un resumen de los pasos necesarios en la preparación de la biblioteca, incluyendo la duración y las paradas opcionales.
| Preparación de la biblioteca | Proceso | Duración | Parada opcional |
|---|---|---|---|
| Etiquetado del ADN con códigos de barras | Tagmentación del ADN con el Rapid Barcoding Kit V14 | 15 minutos | 4 °C durante la noche |
| Agrupación y purificación de muestras | Agrupar las bibliotecas y purificarlas con microesferas AMPure XP | 25 minutos | 4 °C durante la noche |
| Unión del adaptador rápido | Unir los adaptadores de secuenciación a los extremos de ADN | 5 minutos | Recomendamos vivamente secuenciar la biblioteca tan pronto como se hayan incorporado los adaptadores |
| Preparación y carga de la celda de flujo | Acondicionar la celda de flujo y cargar la biblioteca | 10 minutos |
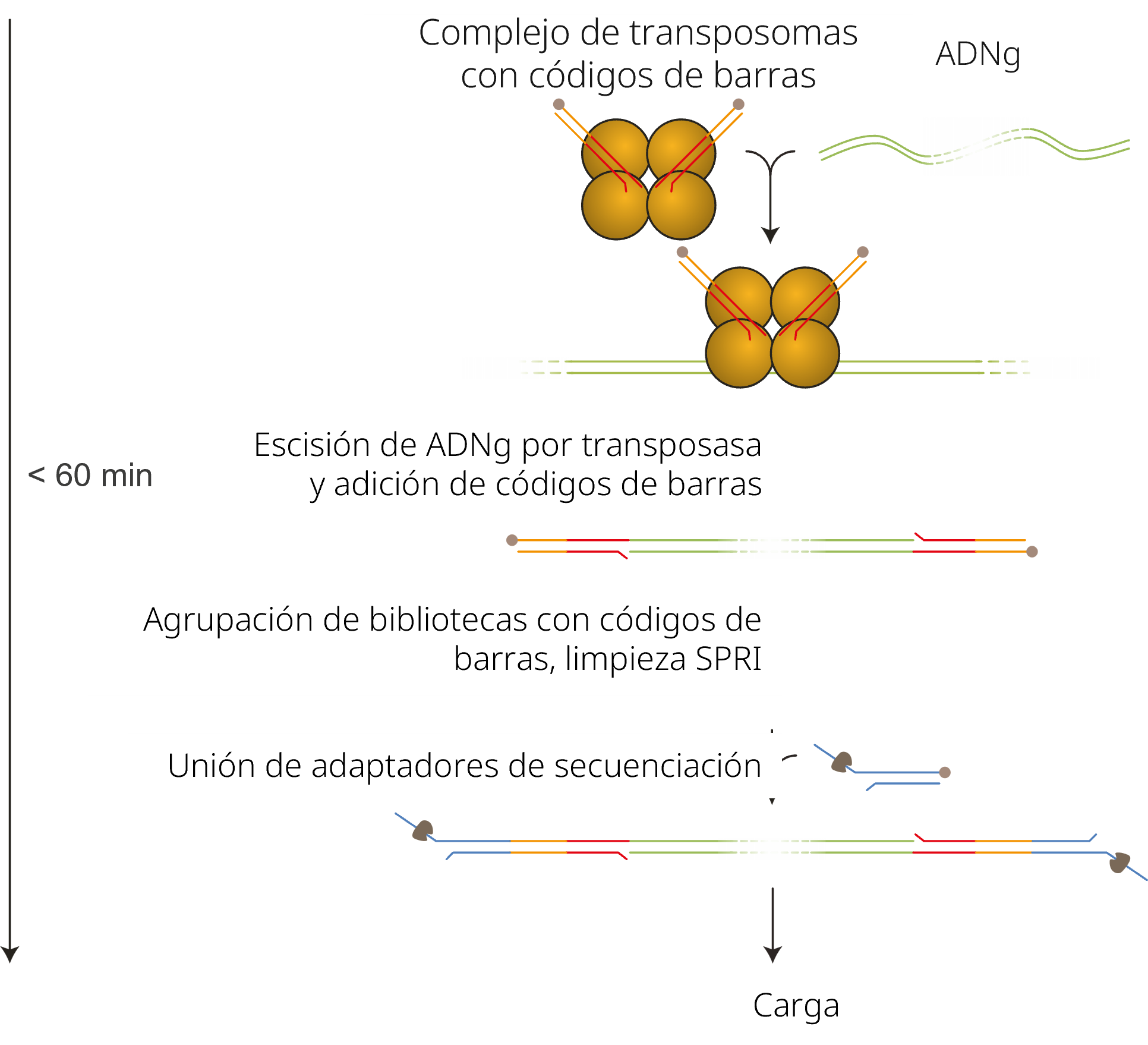
Secuenciación y análisis
Pasos:
- Iniciar un experimento de secuenciación con el software MinKNOW, que recopilará datos sin procesar, identificará las bases y demultiplexará las lecturas con código de barras.
- Realizar el análisis de datos en EPI2ME mediante el modo isolate del flujo de trabajo wf-bacterial-genomes.
Compatibilidad de este protocolo
Este protocolo debe utilizarse solo con:
- Kit Rapid Barcoding 24 V14 (SQK-RBK114.24)
- Kit Rapid Barcoding 96 V14 (SQK-RBK114.96)
- Celdas de flujo R10.4.1 (FLO-MIN114)
- Kit Flow Cell Wash Kit (EXP-WSH004)
- Kit Flow Cell Priming Kit V14 (EXP-FLP004)
- Sequencing Auxiliary Vials V14 (EXP-AUX003)
- Rapid Adapter Auxiliary V14 (EXP-RAA114)
- MinION Mk1D - Requisitos informáticos del MinION Mk1D
2. Equipos y consumibles
Material
- 200 ng of extracted gDNA per sample
- Uno de los siguientes kits: Rapid Barcoding Kit 24 V14 (SQK-RBK114.24) o Rapid Barcoding Kit 96 V14 (SQK-RBK114.96)
Consumibles
- MinION/GridION Flow Cell
- MagMAX™ DNA Multi-Sample Ultra 2.0 Kit (ThermoFisher, cat # A36570)
- MagMAX™ DNA Cell and Tissue Extraction Buffer (ThermoFisher, cat # A45469)
- Kit Qubit 1x dsDNA HS (ThermoFisher, Q33230)
- Qubit 1x dsDNA BR Assay Kit (ThermoFisher, Q33265)
- PowerBead Pro tube (Qiagen, 19301)
- BashingBead Buffer (Zymo, D6001-3-40)
- Phosphate-buffered saline (PBS), pH 7.4 (Thermo Fisher, 10010023)
- TE buffer (Sigma, 8890-100ML)
- 5 M sodium chloride solution (Sigma, S6546)
- CTAB buffer (Promega, MC1411)
- (Optional) Extra reagents for custom CTAB buffer:
- CTAB (Sigma, H6269)
- 0.5 M EDTA (Fisher Scientific, 11568896)
- Trizma® hydrochloride solution (Sigma, T2819)
- Extra reagents for bacteria gDNA extraction:
- Lysozyme human (Sigma, L1667)
- Sodium dodecyl sulfate (SDS) at 10% v/v (Sigma, 71736)
- Achromopeptidase (Sigma, A3547)
- Extra reagents and consumables for hard to lyse organisms gDNA extraction:
- Empty FastPrep® 2mL Lysing Matrix tubes (MP Biomedicals, 115076200)
- Screw Cap for 2 mL Lysing Matrix tubes (MP Biomedicals, 115067005)
- Glass beads, 4 mm (MP Biomedicals, 116914801)
- Extra reagents for fungi gDNA extraction:
- MetaPolyzyme (Sigma, MAC4L-5MG)
- Agencourt AMPure XP beads (Beckman Coulter, A63881)
- Extra reagents for automated bead-beating gDNA extraction:
- RNase A (QIAGEN, 19101)
- Proteinase K (QIAGEN, 19131)
- Maxwell® RSC PureFood Pathogen kit (Promega, AS1660)
- Seroalbúmina bovina (BSA) (50 mg/ml) (p. ej., Invitrogen™ UltraPure™ BSA 50 mg/ml, AM2616)
- Etanol al 80 % recién preparado con agua sin nucleasas
- Nuclease-free water (e.g. Thermo Scientific, AM9937)
- PCR plate seals
- 96-well PCR plate, semi-skirted (e.g. Starlab, I1402-9800)
- 1.5 ml Eppendorf DNA LoBind tubes
- Tubos Eppendorf DNA LoBind de 2 ml
- 0.2 ml thin-walled PCR tubes or 0.2 ml 96-well PCR plate
- Qubit™ Assay Tubes (Invitrogen, Q32856)
Instrumental
- MinION or GridION device
- MinION/GridION Flow Cell Light Shield
- HulaMixer™ (gentle rotator mixer)
- Vortex Adapter (Qiagen 13000-V1-24)
- Vortex mixer
- Thermal cycler
- Timer
- Thermomixer
- Gradilla magnética
- Centrifugadora de microplacas
- Centrifugadora Eppendorf 5424 (o equivalente)
- Qubit™ fluorometer (or equivalent for QC check)
- Multichannel pipette and tips
- P1000 pipette and tips
- P200 pipette and tips
- P100 pipette and tips
- P20 pipette and tips
- P2 pipette and tips
- Ice bucket with ice
La anterior lista de materiales, consumibles e instrumental corresponde a todos los métodos de extracción de la sección Preparación de muestras, así como de la sección Preparación de la biblioteca. Solo serán necesarios los consumibles correspondientes al método de extracción seleccionado, de acuerdo a su tipo de muestra y los incluidos en la sección Preparación de la biblioteca.
En este protocolo, se necesitan las siguientes cantidades por muestra:
Requisitos de cantidad por muestra según el método de extracción:
Extracción general de ADN genómico mediante homogeneización con microesferas: 1 ml de cultivo líquido incubado durante la noche (~1 × 10⁸–10⁹ UFC/ml) o media asa de colonias procedentes de una placa
Extracción de ADN genómico bacteriano: 200 µl de cultivo líquido incubado durante la noche (~1 × 10⁸–10⁹ UFC/ml) o 1/8 de un asa de colonias procedentes de una placa
Extracción de ADN genómico en organismos difíciles de lisar: 5 – 10 mg de células de medios sólidos o líquidos
Extracción de ADN genómico de hongos: 2 ml de cultivo incubado durante la noche con una concentración aproximada de ~1 × 10⁷ UFC/ml o un asa de siembra de 10 µl de colonias procedentes de una placa
En la preparación de la biblioteca, se requieren 200 ng en 10 µl de ADN genómico extraído por muestra.
Reactivos de otros fabricantes
Según el protocolo de extracción que se utilice, no serán necesarios todos los reactivos de otros fabricantes.
Hemos validado y recomendamos el uso de los reactivos de otros fabricantes utilizados en este protocolo. Oxford Nanopore Technologies no ha probado otras alternativas.
Respecto a los reactivos de otros fabricantes, recomendamos seguir las instrucciones del fabricante.
Tampón CTAB personalizado
Es posible preparar un tampón CTAB personalizado en lugar de comprarlo. A continuación se indican los reactivos y las concentraciones necesarios, con ejemplos sugeridos que incluyen un margen adicional.
| Reactivo | Existencias | Concentración final | Volumen 12 muestras | Volumen 24 muestras |
|---|---|---|---|---|
| CTAB | - | 2 % v/v | 60 µl | 120 µl |
| EDTA | 0,5 M | 40 mM | 240 µl | 480 µl |
| Cloruro de sodio | 5 M | 1,4 M | 833 µl | 1 666 µl |
| Solución de clorhidrato de Trizma, pH 8 | 1 M | 100 mM | 300 µl | 600 µl |
| Agua libre de nucleasas | - | - | 1 567 µl | 3 134 µl |
| Total | - | - | 3 000 µl | 6 000 µl |
En muestras estafilocócicas
El tampón de lisis estafilocócico (SLB) es necesario en el método de extracción de ADNg bacteriano con muestras de Staphylococcus.
| Reactivo | Existencias | Concentración final | Volumen (12 muestras con excedente) | Volumen (24 muestras con excedente) |
|---|---|---|---|---|
| Solución de clorhidrato de Trizma, pH 9 | 1 M | 100 mM | 150 ul | 300 µl |
| Cloruro de sodio | 5 M | 10 mM | 3 ul | 6 µl |
| SDS | 10 % v/v | 0,1 % v/v | 15 ul | 30 µl |
| Agua sin nucleasas | - | - | 1 332 ul | 2 664 µl |
| Volumen total | - | - | 1 500 ul | 3 000 µl |
El SDS del tampón de lisis estafilocócico (SLB) es esencial en la prevención de la degradación del ADN de los estafilococos. Excluir el SDS del tampón da como resultado una mayor difuminación del ADN cuando se ejecuta en un gel.
ADN inicial
Control de calidad del ADN inicial
Es importante que el ADN inicial cumpla con los requisitos de cantidad y calidad. Usar demasiado ADN, poco o de mala calidad (p. ej., que esté muy fragmentado, que contenga ARN o contaminantes químicos), puede afectar a la preparación de la biblioteca.
Encontrará instrucciones sobre cómo realizar el control de calidad de la muestra de ADN en el Protocolo de control de calidad del ADN/ARN inicial.
Contaminantes químicos
Según el método empleado para extraer el ADN de la muestra original, es posible que ciertos contaminantes químicos permanezcan en el ADN purificado, lo que afecta a la eficiencia de la preparación de la biblioteca y a la calidad de la secuenciación. Encontrará más información sobre contaminantes en la página Contaminantes de la comunidad.
Comprobación de la celda de flujo
Recomendamos comprobar el número de poros de la celda de flujo antes de iniciar un experimento de secuenciación. Esto debe hacerse en un plazo de 12 semanas desde la compra de celdas de flujo MinION, GridION o PromethION o en un plazo de cuatro semanas desde la compra de celdas de flujo Flongle. Oxford Nanopore Technologies sustituirá cualquier celda de flujo con un número de poros inferior al indicado en la tabla siguiente, siempre que el resultado se comunique en un plazo de dos días tras realizar la comprobación de la celda de flujo y se hayan seguido las recomendaciones de almacenamiento. Realizar la comprobación del flow cell siguiendo las instrucciones del documento Flow Cell Check.
| Celdas de flujo | Número mínimo de poros activos cubiertos por la garantía |
|---|---|
| MinION/GridION | 800 |
| PromethION | 5 000 |
El adaptador rápido (RA) utilizado en este kit y protocolo no es intercambiable con otros adaptadores de secuenciación.
Contenido del kit Rapid Barcoding 24 V14 (SQK-RBK114.24)
Estamos cambiando el fomato de los códigos de barras incluidos en este kit por un formato en placa., lo que reducirá la producción de residuos plásticos y favorecerá el uso en aplicaciones automatizadas.
Formato en placa
| Nombre | Acrónimo | Color del tapón | Número de viales | Volumen por vial (µl) |
|---|---|---|---|---|
| Rapid Adapter | RA | Verde | 1 | 15 |
| TAdapter Buffer | ADB | Transparente | 1 | 100 |
| AMPure XP Beads | AXP | Ámbar | 2 | 1200 |
| Elution Buffer | EB | Negro | 1 | 500 |
| Sequencing Buffer | SB | Rojo | 1 | 700 |
| Library Beads | LIB | Rosa | 1 | 600 |
| Library Solution | Tapón blanco, etiqueta rosa | 1 | 600 | |
| Flow Cell Flush | FCF | Tapón transparente, etiqueta azul claro | 1 | 8000 |
| Flow Cell Tether | FCT | Morado | 1 | 200 |
| Placa de códigos de barras rápidos | RB01-24 | - | 2 placas, 3 juegos de códigos de barras por placa | 5 µl por pocillo |
Este producto contiene reactivo AMPure XP fabricado por Beckman Coulter, Inc. y puede almacenarse a -20 °C con el kit sin que ello afecte a la estabilidad del reactivo.
Formato del vial
| Nombre | Sigla | Color del tapón | N.º de viales | Volumen por vial (µl) |
|---|---|---|---|---|
| Rapid Adapter | RA | Verde | 1 | 15 |
| Adapter Buffer | ADB | Transparente | 1 | 100 |
| Microesferas AMPure XP | AXP | Ámbar | 2 | 1 200 |
| Elution Buffer | EB | Negro | 1 | 500 |
| Sequencing Buffer | SB | Rojo | 1 | 700 |
| Library Beads | LIB | Rosa | 1 | 600 |
| Library Solution | LIS | Tapón blanco, etiqueta rosa | 1 | 600 |
| Flow Cell Flush | FCF | Azul | 6 | 1 170 |
| Flow Cell Tether | FCT | Violeta | 1 | 200 |
| Códigos de barras rápidos | RB01-24 | Transparente | 24 | 15 |
Este producto contiene reactivo AMPure XP, fabricado por Beckman Coulter, Inc. y puede almacenarse a -20 °C con el kit sin que ello afecte a la estabilidad del reactivo.
Contenido del kit Rapid Barcoding 96 V14 (SQK-RBK114.96)
| Nombre | Sigla | Color del tapón | N.º de viales | Volumen por vial (µl) |
|---|---|---|---|---|
| Rapid Adapter | RA | Verde | 2 | 15 |
| Adapter Buffer | ADB | Transparente | 1 | 100 |
| microesferas AMPure XP | AXP | Ámbar | 3 | 1 200 |
| Elution Buffer | EB | Negro | 1 | 1 500 |
| Sequencing Buffer | SB | Rojo | 1 | 1 700 |
| Library Beads | LIB | Rosa | 1 | 1 800 |
| Library Solution | LIS | Tapón blanco, etiqueta rosa | 1 | 1 800 |
| Flow Cell Flush | FCF | Transparente | 1 | 15 500 |
| Flow Cell Tether | FCT | Morado | 2 | 200 |
| Códigos de barras rápidos | RB01-96 | - | 3 placas | 8 µl por pocillo |
Este producto contiene el reactivo AMPure XP, fabricado por Beckman Coulter, Inc. y puede almacenarse a -20 °C con el kit sin que ello afecte a la estabilidad del reactivo.
3. Selección del método de extracción de muestras
Decisión sobre el método de extracción optimizado
Hemos desarrollado cuatro métodos de extracción optimizados para generar ADN genómico de gran calidad a partir de sus cultivos celulares, lo que permite maximizar el rendimiento de la secuenciación utilizando este método.
¿Qué método de extracción es el adecuado para mí?
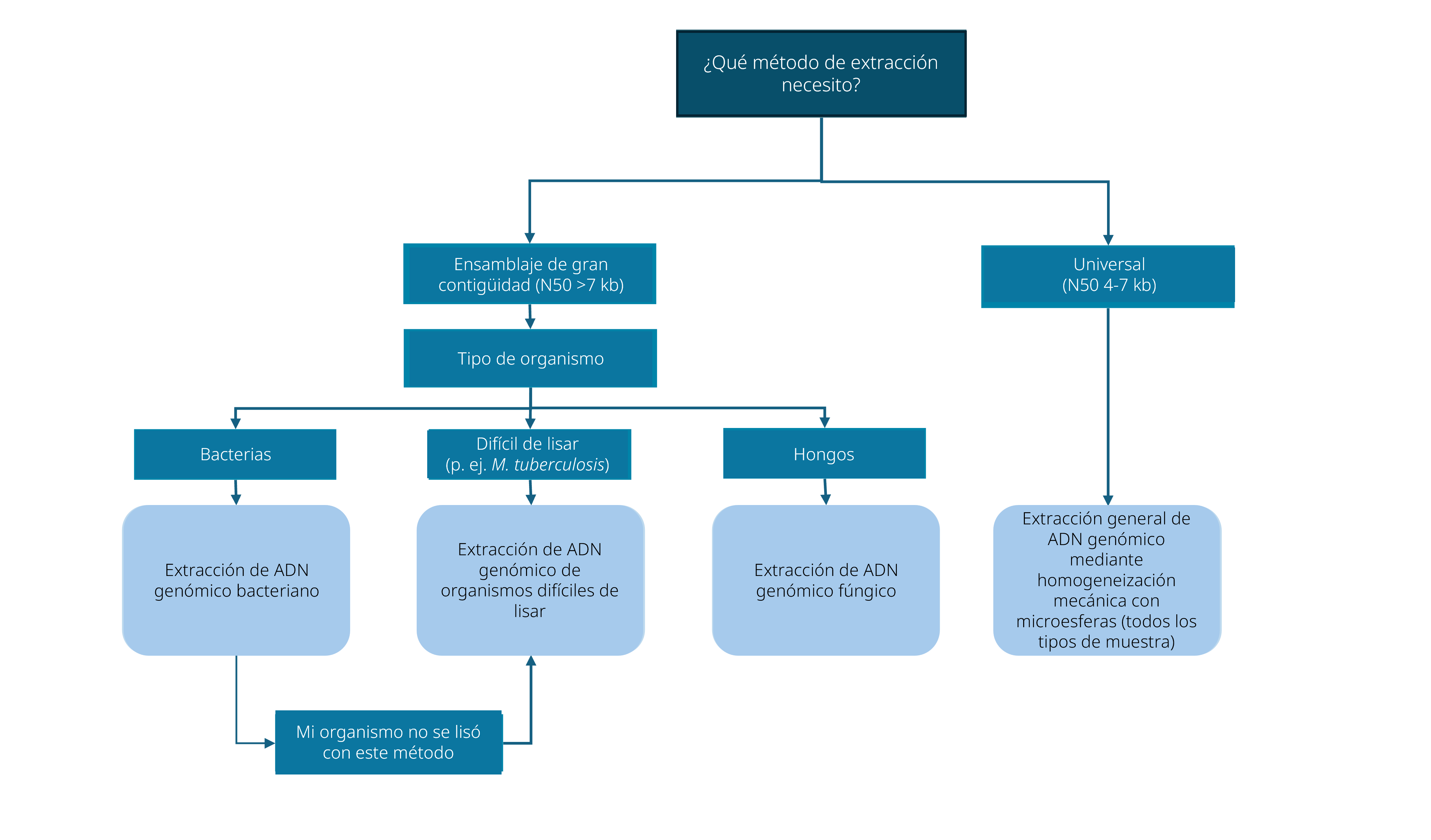
| Método de extracción de muestras | Tipo de muestra | Muestra inicial | Rendimiento esperado | Número de integridad del ADN (DIN) esperado | Longitud media de lecturas de secuenciación | Kits de extracción utilizados |
|---|---|---|---|---|---|---|
| Extracción general de ADN genómico mediante homogeneización con microesferas | Aplicaciones generales: bacterias, hongos o levaduras | 1 ml de cultivo líquido incubado durante la noche (~1 × 10⁸–10⁹ UFC/ml) o la mitad de un asa de colonias de una placa | >200 ng/µl por muestra | 7-9 | ~4-7 kb | QIAGEN PowerBead Tube y MagMAX™ DNA Multi-Sample Ultra 2.0 Kit |
| Extracción de ADNg bacteriano | Bacteriano | 200 µl de cultivo líquido incubado durante la noche (~1 × 10⁸–10⁹ UFC/ml) o 1/8 de un asa de siembra de una placa | ~15-20 ng/µl por muestra | 9 | >7 kb - El tamaño variará en función de la especie de la cantidad de muestra inicial | Kit de extracción de ADNg genómico NEB Monarch Spin |
| Extracción de ADNg de organismos difíciles de lisar | Mycobacterium tuberculosis (o muestras bacterianas difíciles de extraer) | 5-10 mg de células de medios sólidos o líquidos | ~15-40 ng/µl por muestra | 8 | >7 kb: el tamaño variará en función de la especie de la muestra inicial | Kit NEB Monarch Spin gDNA Extraction |
Nota: El rendimiento, el DIN y la longitud de lectura de secuenciación del ADN extraído pueden variar en función de la calidad de la muestra y la especie. Siga el método correcto y utilice muestras de buena calidad.
Siga los enlaces de la tabla anterior, donde se encuentra la documentación de los métodos de extracción.
También puede encontrar estos métodos de extracción en la pestaña Protocolos de extracción Documentación de la comunidad Nanopore.
4. Preparación de la biblioteca
Material
- 200 ng of extracted gDNA per sample
- Códigos de barras rápidos (RB01-24 o RB01-96)
- Rapid Adapter (RA)
- Adapter Buffer (ADB)
- AMPure XP Beads (AXP)
- Elution Buffer (EB)
Consumibles
- Qubit 1x dsDNA BR Assay Kit (ThermoFisher, Q33265)
- Nuclease-free water (e.g. Thermo Scientific, AM9937)
- Etanol al 80 % recién preparado con agua sin nucleasas
- Placa de PCR Eppendorf twin.tec® LoBind de 96 pocillos, con semifaldón y sellado térmico (Eppendorf™, 0030129504)
- 0.2 ml thin-walled PCR tubes
- 1.5 ml Eppendorf DNA LoBind tubes
- Tubos Eppendorf DNA LoBind de 2 ml
- Qubit™ Assay Tubes (Invitrogen, Q32856)
Instrumental
- Ice bucket with ice
- Timer
- Thermal cycler
- Centrifugadora de microplacas
- Gradilla magnética
- Hula mixer (gentle rotator mixer)
- Qubit™ fluorometer (or equivalent for QC check)
- P1000 pipette and tips
- P200 pipette and tips
- P100 pipette and tips
- P20 pipette and tips
- P10 pipette and tips
- P2 pipette and tips
- Multichannel pipette and tips
Requisitos de uso de códigos de barras rápidos y capacidad de procesamiento de muestras con NO-MISS
Este método se ha desarrollado para procesar simultáneamente 24 muestras con un tamaño de genoma de hasta 7 Mb.
En muestras con un genoma de mayor tamaño (>7 Mb), recomendamos reducir a 8 el número de muestras que se etiqueten con códigos de barras y se secuencien simultáneamente.
A fin de obtener un resultado óptimo, en la actualidad no recomendamos utilizar menos de 4 códigos de barras.
Nota: este método ofrece un proceso estandarizado de procesamiento de muestras que hemos validado internamente. Estos ajustes son aplicables a la mayoría de aplicaciones y recomendamos a los usuarios nuevos que sigan el método recomendado. Los usuarios con experiencia pueden ajustar el rendimiento de las muestras (de 4 a 48 muestras con códigos de barras) en función de la calidad de las muestras, el tamaño del genoma y los requisitos de cobertura.
Verificar la celda de flujo
Recomendamos verificar la celda de flujo antes de preparar la biblioteca y comprobar que tiene suficientes poros para realizar una buena sequenciación.
Consultar las instrucciones de comprobación de la celda de flujo en el protocolo MinKNOW, donde encontrará más información.
Programar el termociclador: a 30 °C durante 2 minutos y a 80 °C durante 2 minutos.
Descongelar los componentes del kit a temperatura ambiente, centrifugar brevemente y mezclar con la pipeta, como se indica en la tabla a continuación:
| Reactivo | 1. Descongelar a temperatura ambiente | 2. Centrifugar brevemente | 3. Mezclar bien con la pipeta |
|---|---|---|---|
| Códigos de barras rápidos (RB01-24 o RB01-96) | No congelados | ✓ | ✓ |
| Rapid Adapter | No congelado | ✓ | ✓ |
| microesferas AMPure XP (AXP) | ✓ | ✓ | Mezclar con la pipeta o en vórtex inmediatamente antes de su uso |
| Elution Buffer (EB) | ✓ | ✓ | ✓ |
| Adapter Buffer (ADB) | ✓ | ✓ | Mezclar en vórtex |
Prepare el ADN en agua sin nucleasas.
- Transferir 200 ng de ADN genómico por muestra a tubos de PCR de pared fina (0,2 ml) o a una placa de PCR Eppendorf twin.tec® PCR 96 LoBind.
- Ajustar el volumen de cada muestra a un total de 10 μl con agua sin nucleasas.
- Mezclar bien el contenido de los tubos con una pipeta y centrifugar brevemente en una microcentrífuga.
Seleccionar un código de barras único para cada muestra que se vaya a secuenciar en la misma celda de flujo.
Nota: utilizar un código de barras por muestra.
Los pocillos de la placa de códigos de barras son de un solo uso. Comprobar que los pocillos asignados estén sellados antes de utilizarlos y desechar una vez abiertos o perforados.
En el tubo de PCR de 0,2 ml o en la placa de PCR Eppendorf twin.tec® LoBind de 96 pocillos, mezclar lo siguiente:
| Reactivo | Volumen por muestra |
|---|---|
| Plantilla de ADN (200 ng del paso anterior) | 10 μl |
| Códigos de barras rápidos (RB01-24 o RB01-96, uno para cada muestra) | 1,5 μl |
| Total | 11,5 μl |
Mezclar con la pipeta hasta homogeneizar bien los componentes y centrifugar brevemente.
Incubar los tubos o la placa a 30 °C durante 2 minutos y a 80 °C durante 2 minutos. Colocar los tubos o la placa a enfriar en hielo.
Centrifugar brevemente los tubos o la placa.
Agrupar las muestras etiquetadas en un tubo de 2 ml Eppendorf DNA Lobind y anotar el volumen total.
| . | Volumen por muestra | 24 muestras |
|---|---|---|
| Volumen total | 11,5 µl | 276 µl |
Resuspender las microesferas AMPure XP Beads (AXP) mediante vórtex.
Añadir un volumen igual de microesferas AMPure XP (AXP) resuspendidas a la muestra agrupada con códigos de barras y mezclar dando golpecitos al tubo.
| . | Volumen por muestra | 24 muestras |
|---|---|---|
| Volumen de microesferas AMPure XP (AXP) añadido | 11,5 µl | 276 µl |
Incubar en un mezclador Hula (mezclador rotatorio) durante 10 minutos a temperatura ambiente.
Preparar al menos 2 ml de etanol al 80 % fresco en agua sin nucleasas.
Centrifugar la muestra brevemente y sedimentar en una gradilla magnética. Sin mover el tubo, retirar el sobrenadante con una pipeta.
Sin mover el tubo, lavar las microesferas con 1 ml de etanol al 80 % recién preparado sin alterar el sedimento. Retirar el etanol con una pipeta y desechar.
Repetir el paso anterior.
Centrifugar el tubo brevemente y colocarlo de nuevo en la gradilla magnética. Retirar con una pipeta cualquier residuo de etanol. Dejar secar el sedimento durante 30 segundos, sin dejar que se agriete.
Quitar el tubo del soporte magnético y resuspender el sedimento en 15 µl de Elution Buffer (EB). Incubar durante 10 minutos a temperatura ambiente.
Sedimentar las microesferas en la gradilla magnética, durante al menos 1 minuto, hasta que el eluido se vuelva claro e incoloro.
Retirar y conservar todo el volumen de eluido en un tubo Eppendorf DNA LoBind de 1,5 ml.
- Retirar y conservar el eluido, que contiene la biblioteca de ADN, en un tubo Eppendorf DNA LoBind de 1,5 ml.
- Desechar el sedimento.
Cuantificar 1 µl de muestra eluida con un fluorímetro Qubit y un kit Qubit ADNbc BR.
De 24 muestras, por lo general se obtienen ~150 ng/µl, suponiendo que se haya conservado el 70 % del ADN durante el lavado.
Transferir 11 µl de la muestra a un tubo Eppendorf DNA LoBind de 1,5 ml.
En un tubo de 1,5 ml Eppendorf DNA LoBind, diluir el Rapid Adapter (RA) como se indica a continuación y mezclar con la pipeta:
| Reactivo | Volumen |
|---|---|
| Rapid Adapter (RA) | 1,5 μl |
| Adapter Buffer (ADB) | 3,5 μl |
| Total | 5 μl |
Añadir 1 µl del Rapid Adapter (RA) diluido al ADN etiquetado.
Mezclar golpeando suavemente el tubo con el dedo y centrifugar brevemente.
Incubar la reacción durante 5 minutos a temperatura ambiente.
Nota: durante la incubación, continuar con la preparación y carga de la celda de flujo.
La biblioteca preparada se usará para cargar la celda de flujo. Conservar la biblioteca en hielo hasta el momento de cargar.
5. Acondicionar y cargar la celda de flujo MinION/GridION
Material
- Flow Cell Flush (FCF)
- Flow Cell Tether (FCT)
- Library Beads (LIB)
- Sequencing Buffer (SB)
Consumibles
- MinION/GridION Flow Cell
- Seroalbúmina bovina (BSA) (50 mg/ml) (p. ej., Invitrogen™ UltraPure™ BSA 50 mg/ml, AM2616)
- 1.5 ml Eppendorf DNA LoBind tubes
Instrumental
- MinION or GridION device
- MinION/GridION Flow Cell Light Shield
- P1000 pipette and tips
- P100 pipette and tips
- P20 pipette and tips
- P10 pipette and tips
Nótese, este kit es compatible solo con las celdas de flujo R10.4.1 (FLO-MIN114).
Sacar la celda de flujo de la nevera y dejar a temperatura ambiente durante 20 minutos, mejorará la visibilidad de la matriz durante el acondicionamiento y carga de la muestra.
Preparación y carga de celdas de flujo
Les recomendamos que miren el vídeo Preparar y cargar la celda de flujo antes de realizar el primer experimento.
Descongelar los viales Sequencing Buffer (SB), Library Beads (LIB) o Library Solution (LIS), -si se requiere-, Flow Cell Tether (FCT) y un tubo de Flow Cell Flush (FCF) a temperatura ambiente y mezclar en vórtex. Centrifugar brevemente y colocar en hielo.
Añadir seroalbúmina bovina (BSA) en una concentración total de 0,2 mg/ml a la mezcla de acondicionamiento. De este modo se obtendrá un rendimiento de secuenciación óptimo y mejorará la eficiencia de las celdas de flujo R10.4.1 (FLO-MIN114).
Nota: no recomendamos ningún otro tipo de albúmina (por ejemplo, albúmina sérica humana recombinante).
Preparar la mezcla de acondicionamiento con BSA, añadiendo los siguientes reactivos a un tubo Eppendorf DNA LoBind de 1,5 ml nuevo. Invertir el tubo varias veces y mezclar con la pipeta a temperatura ambiente:
| Reactivos | Volumen por celda de flujo |
|---|---|
| Flow Cell Flush (FCF) | 1 170 µl |
| Seroalbúmina bovina (BSA) a 50 mg/ml | 5 µl |
| Flow Cell Tether (FCT) | 30 µl |
| Volumen total | 1 205 µl |
Abrir la tapa del dispositivo MinION o GridION y deslizar la celda de flujo debajo del clip. Presionar firmemente la tapa del puerto de purgado hasta lograr un contacto térmico y eléctrico adecuados.

Antes de cargar la biblioteca, realizar una evaluación de la celda de flujo para determinar el número de poros disponible.
Este paso se puede omitir si se ha evaluado la celda de flujo con anterioridad.
Encontrará más información en las instrucciones de comprobación de la celda de flujo del protocolo de MinKNOW.
Deslizar la cubierta del puerto de purgado de la celda de flujo en el sentido de las agujas del reloj.

Tenga cuidado al extraer el tampón de la celda de flujo. No extraer más de 20-30 µl y comprobar que la matriz de poros esté cubierta de tampón en todo momento. Introducir burbujas de aire en la matriz es capaz de dañar los poros de forma irreversible.
Tras abrir el puerto de purgado, comprobar si hay burbujas de aire bajo la tapa. Extraer una pequeña cantidad de volumen hasta eliminar cualquier burbuja:
- Ajustar una pipeta P1000 a 200 µl.
- Insertar la punta en el puerto de purgado.
- Girar la rueda hasta que el dial muestre 220-230 µl o hasta que se vea un pequeño volumen de tampón entrar en la punta de la pipeta.
Nota: comprobar que el tampón fluya desde el puerto de purgado hasta la matriz de sensores.

Cargar 800 µl de la mezcla por el puerto de purgado, evitando introducir burbujas de aire. Esperar cinco minutos. Mientras tanto, preparar la biblioteca siguiendo los pasos indicados a continuación.

Mezclar minuciosamente con la pipeta el contenido del vial Library Beads (LIB).
El vial Library Beads (LIB) contiene microesferas en suspensión. Esas microesferas sedimentan muy rápido; por eso es crucial mezclarlas justo antes de su uso.
Recomendamos utilizar las Library Beads (LIB) en la mayoría de los experimentos de secuenciación. No obstante, el reactivo Library Solution (LIS) está disponible en caso de utilizar bibliotecas más viscosas.
En un tubo de 1,5 ml preparar la biblioteca de la siguiente manera:
| Reactivo | Volumen por celda de flujo |
|---|---|
| Sequencing Buffer (SB) | 37,5 µl |
| Library Beads (LIB) mezcladas inmediatamente antes de su uso, o Library Solution (LIS), si se utiliza | 25,5 µl |
| Biblioteca de ADN | 12 µl |
| Total | 75 µl |
Terminar de acondicionar la celda de flujo:
- Levantar con cuidado la tapa del puerto SpotON.
- Cargar 200 µl de la mezcla de acondicionamiento en el puerto de purgado (no en el puerto de muestras SpotON), con cuidado de no introducir burbujas de aire.

Mezclar la biblioteca suavemente con la pipeta justo antes de cargarla.
Añadir, gota a gota, 75 μl de biblioteca en el puerto SpotON de la celda de flujo. Dejar que cada gota fluya dentro del puerto antes de añadir la siguiente.

Volver a colocar, con cuidado, la tapa del puerto SpotON, asegurándose de que el tapón encaje en el agujero y cerrar el puerto de purgado.


A fin de obtener resultados de secuenciación óptimos, coloque la pantalla protectora sobre la celda de flujo tan pronto como termine de cargar la biblioteca.
Recomendamos dejar la pantalla protectora puesta mientras la biblioteca esté cargada, incluso durante los lavados y recargas. Retirar la pantalla cuando se haya retirado la biblioteca de la celda de flujo.
Colocar la pantalla protectora de la siguiente manera:
Colocar con cuidado el borde delantero de la pantalla protectora contra el clip. Nota: no forzar la pieza para que pase por debajo.
Posar la pantalla protectora sobre la celda de flujo. La pantalla protectora debería asentarse alrededor de la tapa SpotON y cubrir por completo la sección superior de la celda de flujo.

La pantalla protectora no queda sujeta a la celda de flujo MinION. Tras su colocación, conviene manipular la celda con cuidado.
Cerrar la tapa del dispositivo y configurar un experimento de secuenciación en MinKNOW.
When a flow cell is inserted into the MinION Mk1D, the device lid will sit on top of the flow cell, leaving a small gap around the sides. This is normal and has no impact on the performance of the device.
Please refer to this FAQ regarding the device lid.

6. Adquisición de datos e identificación de bases
Utilizar siempre la versión más reciente del MinKNOW.
Antes de iniciar una un experimento de secuenciación, recomendamos actualizar MinKNOW. El modelo de identificación de bases v4.3, disponible a partir de Dorado 0.5.0, ofrece una mayor precisión en el ADN bacteriano y está incluido en MinKNOW a partir de la versión 24.02.
Encontrará más información sobre la actualización de MinKNOW en nuestro protocolo MinKNOW.
Cómo empezar a secuenciar
El programa MinKNOW realiza el control del dispositivo de secuenciación, la adquisición de datos y la identificación de bases en tiempo real. Compruebe que MinKNOW esté instalado en su ordenador o dispositivo. Encontrará más instrucciones para configurar un experimento de secuenciación en el protocolo MinKNOW.
Recomendamos configurar un experimento en un MinION o GridION, siguiendo las recomendaciones de identificación de bases y adición de códigos descritas a continuación. El resto de parámetros se pueden dejar con su configuración predeterminada.
Cómo empezar un experimento de secuenciación en MinKNOW:
- Acceder a la página inicial y seleccionar Iniciar la secuenciación.
- Introducir los datos del experimento: nombre, posición de la celda de flujo e identificador de la muestra.
- En la página Kits, seleccionar Rapid Barcoding Kit 24 (SQK-RBK114-24) o Rapid Barcoding Kit 96 (SQK-RBK114-96).
- Mantener las opciones predeterminadas: duración de 72 horas y longitud mínima de lectura de 200 bp.
- Configurar la identificación de bases y la asignación de códigos con estos parámetros:
- Activar la identificación de bases.
- En “Modelos”, pulsar Editar y seleccionar Identificación de bases de gran precisión (HAC) en el menú desplegable.
- Activar la asignación de códigos.
- Mantener el resto de opciones en sus valores predeterminados.
- Pulsar Continuar.
- Definir el formato de salida y filtrar con los siguientes ajustes:
- Seleccionar .POD5 como formato de salida.
- Verificar que .FASTQ figure como formato de lecturas identificadas.
- Activar el filtrado.
- Seleccionar Continuar.
- En la página de revisión, al pulsar Inicio comenzará la secuenciación.
7. Análisis de datos
Análisis posterior a la identificación de bases
Recomendamos realizar el análisis con EPI2ME, ya que facilita el análisis bioinformático y permite a los usuarios realizar flujos de trabajo Nextflow en una aplicación de escritorio. EPI2ME contiene una colección de flujos de trabajo bioinformáticos, seleccionados y gestionados prontamente por expertos en análisis de secuencias de lecturas largas.
Aquí encontrará más información sobre los flujos de trabajo disponibles en EPI2ME, junto con la Guía de inicio rápido, útiles para abordar el primer flujo de trabajo bioinformático.
A fin de obtener un ensamblaje y una alineación precisos y fiables de genomas de aislamientos bacterianos o fúngicos generados con los protocolos incluidos, recomendamos utilizar el flujo de trabajo wf-bacterial-genomes. En esencia, el flujo de trabajo es un flujo bioinformático de ensamblaje eficiente que también pule los genomas con Medaka.
Aunque ejecutar el flujo de trabajo con los parámetros predeterminados producirá ensamblajes genómicos de gran calidad, el modo «Isolates» realizará análisis adicionales diseñados para aumentar la calidad del genoma y facilitar su interpretación en patógenos comunes en los ámbitos clínico y de seguridad alimentaria. Los análisis del modo Isolates incluyen MLST (7 genes), confirmación de especies y predicción de resistencia a los antimicrobianos, además de informes específicos de las muestras.
Nota: también es posible ejecutar este flujo de trabajo desde la línea de comandos. Pero, solo recomendamos esta opción a usuarios experimentados. Encontrará más información, en la página wf-bacterial-genomes de GitHub.
Abrir la aplicación EPI2ME desde el acceso directo del escritorio.
En la página de inicio, abrir la pestaña de flujo de trabajo en la barra lateral izquierda.

Ir a la pestaña Flujos de trabajo disponibles y pulsar la opción wf-bacterial-genomes.

Pulsar «Instalar».

Ir a la pestaña *Instalado* y pulsar en el flujo de trabajo instalado wf-bacterial-genomes.

Si el flujo de trabajo ya estaba instalado, pulsar «Actualizar flujo de trabajo».
Utilizar la versión 1.3.0 del flujo de trabajo, o una más reciente. A fin de obtener los mejores resultados, recomendamos utilizar la última versión de nuestros flujos de trabajo.


Pulsar en Ejecutar este flujo de trabajo abrirá el asistente de inicio.

Configurar el experimento y cargar el archivo FASTQ en las Opciones de entrada. Respecto a los demás parámetros, recomendamos mantener la configuración predeterminada.

Marcar la casilla de verificación «Aislar», que habilitará el modo de aislamiento.

Ir a la pestaña «Configuración de Nextflow» y asignar un «Nombre del experimento» como identificador del análisis.

Pulsar Iniciar el flujo de trabajo.
Comprobar que todas las opciones de los parámetros tengan marcas verdes.

Cuando el flujo de trabajo finalice, se generará un informe.
8. Reutilización y devolución de celdas de flujo
Material
- Kit Flow Cell Wash (EXP-WSH004)
Cuando el experimento de secuenciación haya terminado, si quiere reutilizar la celda de flujo, siga las instrucciones del protocolo Flow Cell Wash Kit y guarde la celda de flujo a entre 2 °C y 8 °C.
El protocolo del kit de lavado de celdas de flujo está disponible en la comunidad Nanopore.
Cuando termine el experimento, recomendamos lavar la celda de flujo lo antes posible. Si no es posible, dejar en el dispositivo y lavar al día siguiente.
Otra opción es seguir el procedimiento de devolución y enviarla a Oxford Nanopore Technologies.
Si encuentra algún problema o tiene dudas sobre su experimento de secuenciación, consulte la sección Resolución de problemas de este protocolo.
9. Problemas durante la extracción de ARN y preparación de la biblioteca
A continuación hay una lista de los problemas más frecuentes, con algunas posibles causas y soluciones propuestas.
También disponemos de una sección de Preguntas frecuentes (FAQ) en la sección Support de la comunidad Nanopore.
Si ha probado las soluciones propuestas y los problemas aún persisten, póngase en contacto con el departamento de Asistencia técnica, bien por correo electrónico (support@nanoporetech.com) o a través del Live Chat de la comunidad Nanopore.
Baja calidad de la muestra
| Observación | Posibles causas | Comentarios y acciones recomendadas |
|---|---|---|
| Lisis ineficaz | La actividad enzimática se ha degradado en la solución o la especie aislada es difícil de lisar. | - Preparar una solución enzimática nueva. - Siga el método de extracción de ADNg difícil de lisar. - Aumentar la incubación de la enzima durante más de 10 minutos. |
| Baja concentración de ADN | Escasa cantidad de muestra inicial en el método de extracción | - Compruebe la cantidad de muestra inicial utilizada - Añada más cantidad de muestra y vuelva a realizar la extracción - Eluir en menos tampón de elución - Concentre el ADN con un lavado de microesferas AMPure XP al 0,4×. |
| Índice de integridad de ADN bajo (DIN) | Mala calidad o poca concentración de la muestra inicial | - Repetir la extracción con una solución enzimática recién preparada. - Concentrar la cantidad de la muestra inicial con un lavado a una proporción de 0,4×. |
| Bajo rendimiento de la secuenciación | Escasa concentración de la muestra. | - Concentrar el ADN con un paso de lavado de microesferas AMPure XP al 0,4× para eliminar posibles inhibidores. - Comprobar la concentración y calidad del ADN. La presencia de ARN puede afectar a la cuantificación del ADN total. |
| Pureza del ADN baja (las lecturas de Nanodrop para el ADN OD 260/280 son <1,8 y OD 260/230 es <2,0-2,2) | El método de extracción de ADN no proporciona la pureza necesaria | Los efectos de los contaminantes se muestran en la sección Contaminantes. Probar un método de extracción alternativo que no dé lugar a la transferencia de contaminantes. Considere la posibilidad de realizar un paso adicional de purificación / lavado SPRI. |
Escasa recuperación de ADN tras la purificación con microesferas AMPure
| Observaciones | Posibles causas | Comentarios y acciones recomendadas |
|---|---|---|
| Escasa recuperación | Pérdida de ADN debido a una proporción de microesferas AMPure por muestra inferior a la prevista | 1. Las microesferas AMPure se sedimentan rápidamente, por lo que deben resuspenderse bien antes de añadirlas a la muestra. 2. Si la proporción de microesferas AMPure por muestra es inferior a 0.4:1, los fragmentos de ADN, sean del tamaño que sean, se perderán durante la limpieza. |
| Escasa recuperación | Los fragmentos de ADN son más cortos de lo esperado | Cuanto menor sea la proporción de microesferas AMPure por muestra, más rigurosa será la selección de fragmentos largos frente a los cortos. Determinar siempre la longitud de la muestra de ADN en un gel de agarosa u otros métodos de electroforesis en gel y, a continuación calcular la cantidad adecuada de microesferas AMPure que se debe utilizar.  |
| Escasa recuperación tras la preparación de los extremos | El paso de lavado utilizó etanol con una concentración inferior al 70 % | El ADN se eluirá de las microesferas cuando se utilice etanol en una concentración inferior al 70 %. Compruebe que utiliza el porcentaje correcto. |
10. Problemas durante el experimento de secuenciación al usar un kit de secuenciación rápida
A continuación hay una lista de los problemas más frecuentes, con algunas posibles causas y soluciones propuestas.
También disponemos de una sección de Preguntas frecuentes (FAQ), en la sección Support de la comunidad Nanopore.
Si ha probado las soluciones propuestas y los problemas aún persisten, póngase en contacto con el departamento de Asistencia técnica, bien por correo electrónico (support@nanoporetech.com) o a través del Live Chat de la comunidad Nanopore.
Menos poros al inicio de la secuenciación que tras la evaluación de la celda de flujo
| Observaciones | Posibles causas | Comentarios y acciones recomendadas |
|---|---|---|
| MinKNOW registró al inicio de la secuenciación un número de poros inferior al número registrado en la evaluación de celda de flujo | Se introdujo una burbuja de aire en la matriz de nanoporos | Tras la evaluación de la celda de flujo, es fundamental eliminar cualquier burbuja de aire del puerto de purgado, antes de acondicionar la celda de flujo. Si no se quitan, pueden desplazarse a la matriz de nanoporos y dañar de manera irreversible los nanoporos expuestos al aire. En este vídeo se muestran algunas buenas prácticas para evitar que esto ocurra. |
| MinKNOW registró al inicio de la secuenciación un número de poros inferior al número registrado en la evaluación de celda de flujo | La celda de flujo no está insertada correctamente en el dispositivo | Detener el ciclo de secuenciación, quitar la celda de flujo del dispositivo e insertarla de nuevo. Comprobar que está firmemente asentada en la base y que ha alcanzado la temperatura deseada. Si procede, probar con una posición diferente en el dispositivo (GridION/PromethION). |
| MinKNOW registró al inicio de la secuenciación un número de poros inferior al número registrado en la evaluación de celda de flujo | Las contaminaciones en la biblioteca dañaron o bloquearon los poros | El número de poros resultante tras la evaluación de la celda de flujo se realiza mediante el control de calidad (QC) de las moléculas de ADN presentes en el tampón de almacenamiento. Al inicio de la secuenciación, se utiliza la misma biblioteca para calcular el número de poros activos. Por este motivo, se estima que puede haber una variabilidad del 10 % en el número de poros detectados. Tener un número de poros considerablemente inferior al inicio de la secuenciación puede deberse a la presencia de contaminantes en la biblioteca que hayan dañado las membranas o bloqueado los poros. Para mejorar la pureza del material de entrada, tal vez sea necesario usar métodos de purificación o extracción de ADN/ARN alternativos. Los efectos de los contaminantes se muestran en la página Contaminants. Probar con un método de extracción alternativo que no provoque el arrastre de contaminantes. |
Error en el script de MinKNOW
| Observaciones | Posibles causas | Comentarios y acciones recomendadas |
|---|---|---|
| MinKNOW muestra el mensaje «Error en el script» | Reiniciar el ordenador y reiniciar MinKNOW. Si el problema persiste, reúna los archivos de registro de MinKNOW y contacte con el departamento de Asistencia técnica. Si no dispone de otro dispositivo de secuenciación, recomendamos que guarde la celda de flujo cargada a 4 °C y contacte con el servicio de asistencia técnica para recibir instrucciones de almacenamiento adicionales. |
Ocupación de poro inferior al 40 %
| Observaciones | Posibles causas | Comentarios y acciones recomendadas |
|---|---|---|
| Ocupación de poros inferior al 40 % | No se ha cargado suficiente biblioteca en la celda de flujo | Compruebe que se carga la concentración correcta de biblioteca de buena calidad en la celda de flujo MinION/GridION. Comprobar la concentración en el protocolo de preparación de bibliotecas. Cuantificar la biblioteca antes de cargarla y calcular los moles con herramientas como la calculadora Promega Biomath, y seleccionar «ADNbc: µg to pmol» |
| Ocupación de poros cercana a 0 | Se utilizó uno de los kits Rapid Sequencing V14 o kit Rapid Barcoding V14 y los adaptadores de secuenciación no se ligaron al ADN | Siga estrictamente el protocolo y utilice los volúmenes y las temperaturas de incubación correctos. También es posible preparar una biblioteca de lambda control para valorar la integridad de los reactivos. |
| Ocupación de poros cercana a 0 | No hay anclajes en la celda de flujo | Los anclajes se añaden durante el acondicionamiento de la celda de flujo (tubo FCT). Añadir FCT (Flow Cell Tether) al tubo de FCF (Flow Cell Flush) antes de acondicionar la celda. |
Longitud de lectura más corta de lo esperado
| Observaciones | Posibles causas | Comentarios y acciones recomendados |
|---|---|---|
| Longitud de lectura más corta de lo esperado | Fragmentación no deseada de la muestra de ADN | La longitud de lectura refleja la longitud del fragmento del ADN introducido. Es posible que la muestra de ADN se fragmente durante la extracción y la preparación de la biblioteca. 1. Revise los métodos de extracción en la comunidad Nanopore donde se describen las prácticas de extracción recomendadas. 2. Visualizar la distribución de la longitud de los fragmentos de las muestras de ADN en un gel de agarosa antes de proceder a la preparación de la biblioteca.  En la imagen anterior, la muestra 1 presenta un peso molecular elevado, mientras que la muestra 2 ha sido sometida a fragmentación. En la imagen anterior, la muestra 1 presenta un peso molecular elevado, mientras que la muestra 2 ha sido sometida a fragmentación. 3. Durante la preparación de la biblioteca, evitar pipetear y agitar en vórtex al mezclar los reactivos. Dar suaves golpes con el dedo o invertir el vial es suficiente. |
Gran proporción de poros no disponibles
| Observación | Posibles causas | Comentarios y acciones recomendadas |
|---|---|---|
Gran proporción de poros no disponibles (mostrados en azul en el panel de canales y en el gráfico de actividad de poros) 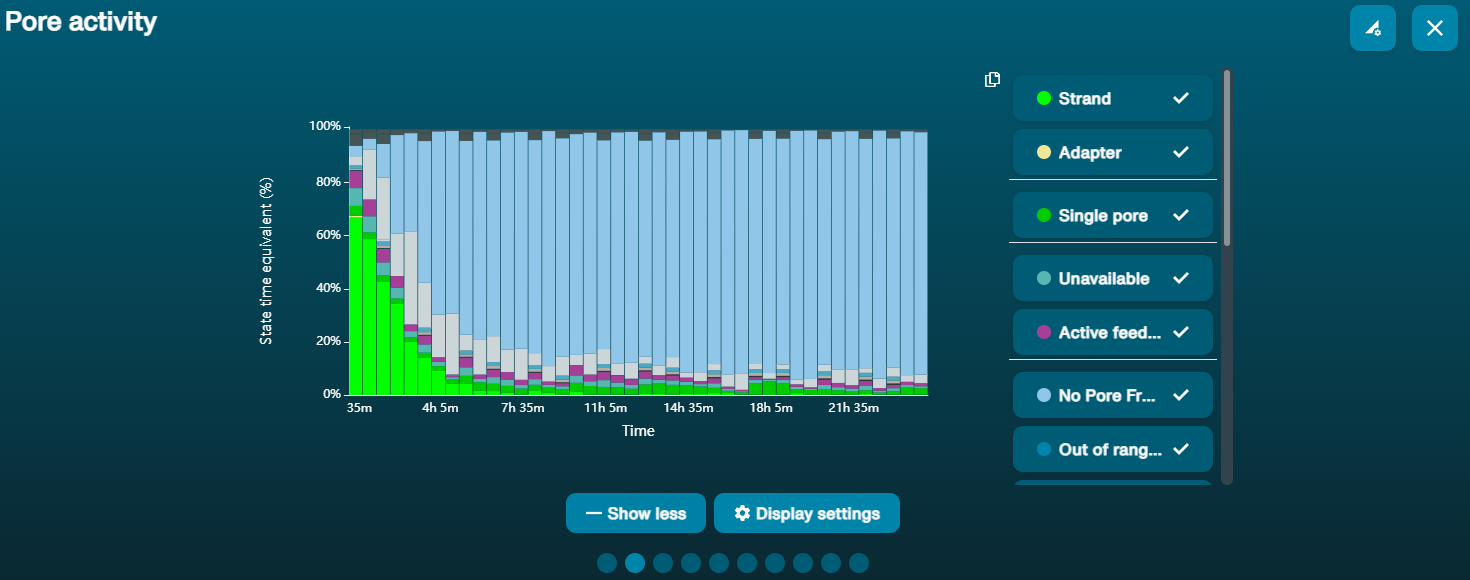 El gráfico de actividad de poros anterior muestra un aumento de la proporción de poros «no disponibles» con el paso del tiempo. El gráfico de actividad de poros anterior muestra un aumento de la proporción de poros «no disponibles» con el paso del tiempo. | Hay contaminantes presentes en la muestra | Es posible eliminar algunos de los contaminantes de los poros mediante la función de desbloqueo incorporada en MinKNOW. Si funciona, el estado de los poros cambiará a "sequencing pores"(poros activos). Si la proporción de poros no disponibles sigue siendo grande o aumenta: 1. Realizar un lavado de nucleasa con el kit Flow Cell Wash Kit (EXP-WSH004), o bien 2. Realizar varios ciclos de PCR e intentar diluir cualquier contaminante que pueda estar causando problemas. |
Gran proporción de poros inactivos
| Observación | Posibles causas | Comentarios y acciones recomendadas |
|---|---|---|
| Gran proporción de poros inactivos o no disponibles (se muestran en azul claro en el panel de canales y en el gráfico de actividad de poros). Los poros o las membranas están irreversiblemente dañados) | Se han introducido burbujas de aire en la celda de flujo | Es posible que las burbujas de aire introducidas durante el acondicionamiento y carga de la celda de flujo hayan dañado los poros de manera irreversible. Ver el vídeo Acondicionamiento y carga de la celda de flujo para evitar que esto ocurra. |
| Gran proporción de poros inactivos o no disponibles | Ciertos compuestos copurificados con el ADN | Compuestos conocidos, incluidos los polisacáridos, que se asocian típicamente con el ADN genómico de las plantas. 1. Consultar el Método de extracción de ADN de hojas vegetales. 2. Purificar con el kit PowerClean Pro de QIAGEN 3. Realizar una amplificación del genoma completo con la muestra original de ADNg con el kit REPLI-g de QIAGEN. |
| Gran proporción de poros inactivos o no disponibles | Hay contaminantes en la muestra | Los efectos de los contaminantes se muestran la página Contaminantes. Probar con un método de extracción alternativo que no provoque el arrastre de contaminantes. |
Fluctuación de la temperatura
| Observación | Posibles causas | Comentarios y acciones recomendadas |
|---|---|---|
| Fluctuación de temperatura | La celda de flujo ha perdido contacto con el dispositivo | Compruebe que haya una almohadilla térmica enganchada en la placa metálica de la parte posterior de la celda de flujo. Reinsertar la celda de flujo y presionar hasta que las clavijas del conector queden bien encajadas en el dispositivo. Si el problema persiste, contacte con el servicio de Asistencia técnica. |
Error al intentar alcanzar la temperatura deseada
| Observación | Posibles causas | Comentarios y acciones recomendadas |
|---|---|---|
| MinKNOW muestra el mensaje "No se ha alcanzado la temperatura deseada" | El instrumento se colocó en un lugar más frío de lo normal o en un lugar con poca ventilación (lo que provoca el sobrecalentamiento de las celdas de flujo). | MinKNOW tiene un tiempo predeterminado para que la celda de flujo alcance la temperatura deseada. Una vez acabado el tiempo, aparece un mensaje de error, pero el experimento de secuenciación continúa. Secuenciar a una temperatura incorrecta puede llevar a una disminución del rendimiento y a generar índices de calidad Qscore menores. Corregir la ubicación del dispositivo, de forma que esté a temperatura ambiente y con buena ventilación; a continuación, reiniciar el proceso en MinKNOW. Encontrará más información sobre el control de temperatura del MinION, en este enlace. |







