Secuenciación rápida de ADN - Kit de 24 códigos de barras 16S V14 (SQK- 16S114.24) (16S_9199_v114_revI_10Feb2026)
MinION: Protocol
Secuenciación rápida de ADN - Kit de 24 códigos de barras 16S V14 (SQK- 16S114.24) V 16S_9199_v114_revI_10Feb2026
Este protocolo describe la amplificación del gen que codifica el ARN ribosómico 16S a partir de ADN genómico.
- Identificación bacteriana a nivel de género
- Multiplexación de hasta 24 muestras diferentes
- Compatible solo con las celdas de flujo R10.4.1
Para uso exclusivo en investigación
FOR RESEARCH USE ONLY
Contents
Introducción
Preparación de la biblioteca
Adquisición de datos e identificación de bases
- 5. Adquisición de datos e identificación de bases
- 6. Reutilización y devolución de celdas de flujo
- 7. Análisis posterior
Resolución de problemas
Descripción general
Este protocolo describe la amplificación del gen que codifica el ARN ribosómico 16S a partir de ADN genómico.
- Identificación bacteriana a nivel de género
- Multiplexación de hasta 24 muestras diferentes
- Compatible solo con las celdas de flujo R10.4.1
Para uso exclusivo en investigación
1. Aspectos generales
Considera probar nuestro nuevo Kit Microbial Amplicon Barcoding 24 V14 (SQK-MAB114.24) para secuenciar sus muestras 16S y ITS.
Las características del Kit Microbial Amplicon Barcoding 24 V14 (SQK-MAB114.24) incluyen:
- Ha mejorado la detección de especies en comparación con el kit SQK-16S114.24, gracias a los cebadores 16S actualizados.
- Incluye identificación de especies de hongos gracias a la inclusión de nuevos cebadores ITS.
Puede encontrar el Kit Microbial Amplicon Barcoding 24 V14 (SQK-MAB114.24) en la tienda online.
Presentación del kit de 24 códigos de barras 16S V14 —16S Barcoding Kit 24 V14— (en adelante nombrado solo en inglés)
Este protocolo describe cómo llevar a cabo la adición de códigos con química rápida de amplicones 16S con el kit 16S Rapid Barcoding Kit 24 V14 (SQK-16S114.24). Dada a la presencia tanto de variantes de regiones altamente conservadas (adecuadas para cebadores universales y señales filogenéticas) como de regiones altamente variables (diferentes en las especies), el gen 16S ARNr se utiliza a menudo para la identificación bacteriana basada en secuencias.
El kit 16S Barcoding Kit 24 V14 permite acceder a una secuenciación rápida del 16S como método de identificación de organismos. Al acotar a una región de interés específica, es posible ver todos los organismos de la muestra sin necesidad de secuenciar regiones innecesarias del genoma, lo que ágiliza la prueba y la hace más barata. Hay 24 códigos diferentes, que permiten agrupar hasta 24 muestras diferentes en un único experimento de secuenciación.
Tras la secuenciación, es posible realizar el análisis utilizando el flujo de trabajo 16S de EPI2ME, que permite clasificar amplicones 16S a partir de las muestras.
Pasos del proceso de secuenciación:
Preparación del experimento
Pasos:
- Extraer el ADNg, evaluar su longitud, cantidad y pureza, según el protocolo de control de calidad Input DNA/RNA QC. Los controles de calidad realizados durante el protocolo son esenciales para garantizar el éxito del experimento.
- Tener a disposición el kit de secuenciación, el instrumental pertinente y los reactivos de otros fabricantes.
- Descargar el programa para obtener y analizar los datos
- Comprobar que la celda de flujo tiene poros suficientes para realizar una buena secuenciación.
Preparación de la biblioteca
La tabla siguiente es un resumen de los pasos necesarios en la preparación de la biblioteca.
| Preparación de la biblioteca | Proceso | Tiempo | Parada opcional |
|---|---|---|---|
| Amplificación por PCR de código 16S | Amplificar el gen 16S utilizando los códigos suministrados en el kit | 10 minutos + PCR | 4 °C durante la noche |
| Agrupación de muestras codificadas y lavado de microesferas | Cuantificar y agrupar las muestras codificadas y realizar un lavado de la biblioteca mediante microesferas | 15 minutos | Almacenamiento a corto plazo o durante un uso repetido a 4 °C, como la recarga de la celda de flujo. Almacenamiento a largo plazo de un solo uso a -80 °C. |
| Ligación de los adaptadores | Ligar los adaptadores de secuenciación rápida a los extremos de ADN | 5 minutos | Recomendamos secuenciar la biblioteca tan pronto como se liguen los adaptadores. |
| Acondicionar y cargar la celda de flujo | Acondicionar la celda de flujo y cargar la biblioteca de ADN | 5 minutos |

Secuenciación y análisis
Pasos:
- Empezar el proceso de secuenciación usando el programa MinKNOW, que obtendrá datos brutos del dispositivo y los convertirá en lecturas de bases identificadas.
- Opcional: Iniciar el programa EPI2ME y seleccionar el flujo de trabajo 16S.
Compatibilidad del protocolo
Este protocolo debería usarse solo en combinación con los siguientes productos:
- 16S Barcoding Kit 24 V14 (SQK-16S114.24)
- Celdas de flujo R10.4.1 (FLO-MIN114)
- Flow Cell Wash Kit (EXP-WSH004)
- Rapid Adapter Auxiliary V14 (EXP-RAA114)
- Sequencing Auxiliary Vials V14 (EXP-AUX003)
- Flow Cell Priming Kit V14 (EXP-FLP004)
- MinION Mk1D - Requisitos informáticos del MinION Mk1D
2. Material y consumibles
Material
- 10 ng de ADN genómico de alto peso molecular
- Kit 16S Barcoding 24 V14 (SQK-16S114.24)
Consumibles
- MinION/GridION Flow Cell
- LongAmp Hot Start Taq 2X Master Mix (NEB, M0533)
- Seroalbúmina bovina (BSA) (50 mg/ml) (p. ej., Invitrogen™ UltraPure™ BSA 50 mg/ml, AM2616)
- Kit Qubit dsDNA HS (Invitrogen Q32851)
- Etanol al 80 % recién preparado con agua sin nucleasas
- Nuclease-free water (e.g. Thermo Scientific, AM9937)
- 1.5 ml Eppendorf DNA LoBind tubes
- Qubit™ Assay Tubes (Invitrogen, Q32856)
- 0.2 ml thin-walled PCR tubes
Instrumental
- MinION or GridION device
- MinION/GridION Flow Cell Light Shield
- Hula mixer (gentle rotator mixer)
- Centrifugadora de microplacas
- Microfuge
- Vortex mixer
- Magnetic separation rack, suitable for 1.5 ml Eppendorf tubes
- Thermal cycler
- P1000 pipette and tips
- P200 pipette and tips
- P100 pipette and tips
- P20 pipette and tips
- P10 pipette and tips
- P2 pipette and tips
- Multichannel pipette and tips
- Ice bucket with ice
- Timer
- Qubit™ fluorometer (or equivalent for QC check)
En este protocolo, necesitará 10 ng de ADN genómico de alto peso molecular por código.
A fin de obtener los mejores resultados, recomendamos utilizar al menos 4 códigos. Si desea multiplexar menos de 4 muestras, procure dividir la(s) muestra(s) entre múltiples códigos, para que al menos se secuencien 4 códigos (por ejemplo, para 2 muestras, utilice cebadores 16S con códigos 01-02 para la muestra A y cebadores 16S con códigos 03-04 para la muestra B). Nótese, la cantidad de muestra requerida por código es de 10 ng de ADNg.
Reactivos de otros fabricantes
Oxford Nanopore Technologies ha probado y recomienda el uso de todos los reactivos de otros fabricantes citados en este protocolo. No se han evaluado otras alternativas.
Recomendamos preparar estos reactivos siguiendo las instrucciones del fabricante.
Verificar la celda de flujo
Antes de empezar el experimento de secuenciación, recomendamos verificar el número de poros disponibles, presentes en la celda de flujo. La comprobación deberá realizarse en las primeras 12 semanas desde su adquisición, si se trata de celdas de flujo MinION/GridION. Oxford Nanopore Technologies sustituirá cualquier celda de flujo con un número de poros inferior al indicado en la tabla siguiente, siempre y cuando el resultado se notifique dentro de los dos días siguientes a la comprobación y se hayan seguido las instrucciones de almacenamiento. Para verificar la celda de flujo, siga las instrucciones del documento Flow Cell Check.
| Celda de flujo | Número mínimo de poros activos cubierto por la garantía |
|---|---|
| MinION/GridION | 800 |
Contenido del Kit de 24 códigos para 16S V14
| Nombre | Sigla | Tapón color | Nº de viales | Volumen (μl) |
|---|---|---|---|---|
| Cebadores 16S con códigos 01-24 | 1-24 | - | 2 placas, 3 sets de códigos por placa | 15 μl por pocillo |
| Rapid Adapter | RA | Verde | 1 | 15 |
| Adapter Buffer | ADB | Transparente | 1 | 100 |
| AMPure XP Beads | AXP | (tapón) Transparente, (etiqueta) Verde azulada | 1 | 6,000 |
| Elution buffer | EB | Negro | 1 | 1 500 |
| EDTA | EDTA | Azul | 1 | 700 |
| Sequencing Buffer | SB | Rojo | 1 | 700 |
| Library Beads | LIB | Rosa | 1 | 600 |
| Library Solution | LIS | (tapón) Blanco, (etiqueta) Rosa | 1 | 600 |
| Flow Cell Flush | FCF | (tapón) Transparente, (etiqueta) Azul claro | 1 | 8,000 |
| Flow Cell Tether | FCT | Violeta | 1 | 200 |
Nota: Este producto contiene un reactivo, AMPure XP, fabricado por Beckman Coulter Inc., que puede conservarse con el kit a -20 °C sin perjudicar su estabilidad.
3. Preparación de la biblioteca
Material
- 10 ng de ADN genómico de alto peso molecular
- Códigos de barras 16S en placa de 96 pocillos, a 1 μM cada uno
- EDTA (ácido etilendiaminotetraacético)
- AMPure XP Beads (AXP)
- Elution Buffer (EB)
- Rapid Adapter (RA)
- Adapter Buffer (ADB)
Consumibles
- LongAmp Hot Start Taq 2X Master Mix (NEB, M0533)
- Kit Qubit dsDNA HS (Invitrogen Q32851)
- Etanol al 80 % recién preparado con agua sin nucleasas
- Nuclease-free water (e.g. Thermo Scientific, AM9937)
- 1.5 ml Eppendorf DNA LoBind tubes
- Qubit™ Assay Tubes (Invitrogen, Q32856)
- 0.2 ml thin-walled PCR tubes
Instrumental
- Centrifugadora de microplacas
- Thermal cycler
- Microfuge
- Hula mixer (gentle rotator mixer)
- Gradilla magnética
- Ice bucket with ice
- Qubit™ fluorometer (or equivalent for QC check)
- P1000 pipette and tips
- P200 pipette and tips
- P100 pipette and tips
- P20 pipette and tips
- P10 pipette and tips
- P2 pipette and tips
- Multichannel pipette and tips
Requisitos mínimos en el uso de cebadores con códigos de barras 16S
A fin de obtener los mejores resultados, recomendamos utilizar al menos 4 códigos de barras. Si desea multiplexar menos de 4 muestras, distribuya la(s) muestra(s) en varios códigos, para que se procesen un mínimo de 4:
- Para procesar 1 muestra, utilice 4 códigos por muestra (por ejemplo, cebadores con códigos 16S 01-04, con 10 ng de muestra A por código de barras).
- Para procesar 2 muestras, utilice dos códigos por muestra, (por ejemplo, cebadores con códigos 16S 01-02 para la muestra A y cebadores con códigos 16S 03-04 para la muestra B).
- Para procesar 3 muestras, ejecute 2 muestras individualmente y una muestra repartida en dos códigos (por ejemplo, cebador con códigos 16S 01 y cebador con códigos 16S 02 para las muestras A y B respectivamente y los cebadores 16S 03-04 para la muestra C).
Nótese, la cantidad de muestra requerida por código es 10 ng de ADNg.
Verificar la celda de flujo
Antes de empezar a preparar la biblioteca, recomendamos verificar la celda de flujo para comprobar que tiene poros suficientes para realizar una buena secuenciación.
Las instrucciones de comprobación de la celda de flujo están disponibles en el protocolo de MinKNOW.
Retirar del congelador una placa de 96 pocillos que contenga los códigos de barras 16S y determinar cuántos se desean utilizar.
Plano de la placa con códigos 16S:
Descongelar los códigos a temperatura ambiente.
Centrifugar brevemente los códigos de barras hasta que el líquido quede en el fondo de los pocillos. Colocar en hielo.
Descongelar la mezcla de reacción LongAmp Hot Start Taq 2X Master Mix, centrifugar brevemente, mezclar bien con la pipeta y colocar en hielo.
Preparar el ADN en agua sin nucleasas.
- Transferir 10 ng de cada muestra de ADN genómico en un tubos de PCR de pared fina (0,2 ml).
- Ajustar el volumen a un total de 15 μl con agua sin nucleasas.
- Dar suaves golpecitos en el tubo con el dedo hasta mezclar bien el contenido, para evitar cizalladuras indeseadas.
- Centrifugar brevemente en la microcentrífuga
En cada tubo de PCR de pared fina (0,2 ml) que contenga una muestra que vaya a ser analizada, preparar la siguiente mezcla:
| Reactivo | Volumen |
|---|---|
| 10 ng de muestra inicial de ADN (del paso anterior) | 15 μl |
| LongAmp Hot Start Taq 2X Master Mix | 25 μl |
| Total | 40 μl |
Nótese, si se cambia la cantidad de muestra inicial, tal vez sea necesario ajustar el número de ciclos de PCR, para obtener el mismo rendimiento.
Mezclar pipeteando con suavidad y centrifugar brevemente la reacción para que los ingredientes se mezclen completamente.
Con ayuda de puntas de pipeta nuevas, perforar con cuidado la superficie de aluminio de los códigos necesarios. Para evitar la contaminación cruzada, utilizar una punta nueva para cada código. Anotar qué números de código se utilizarán en cada muestra.
Los pocillos con los códigos de barras de la placa están diseñados para utilizarse una sola vez. Compruebe que estén sellados antes de utilizarlos y no los reutilice una vez perforados o abiertos.
Con una pipeta multicanal, mezclar los códigos 16S con la pipeta 10 veces. Transferir 10 μl de cada código 16S en los respectivos tubos con muestras.
Mezclar meticulosamente el contenido de los tubos 10 veces con la pipeta y centrifugar brevemente.
Nota: mezclar despacio procurando no introducir burbujas de aire en las reacciones.
Amplificar utilizando las siguientes condiciones de ciclado:
| Fase | Temperatura | Tiempo | Nº de ciclos |
|---|---|---|---|
| Desnaturalización inicial | 95 °C | 1 min | 1 |
| Desnaturalización | 95 °C | 20 s | 25 |
| Hibridación | 55 °C | 30 s | 25 |
| Extensión | 65 °C | 2 min | 25 |
| Extensión final | 65 °C | 5 min | 1 |
| Mantener | 4 °C | ∞ |
Descongelar los componentes del kit a temperatura ambiente, centrifugar brevemente y mezclar con la pipeta, como se indica en la tabla a continuación:
| Reactivo | 1. Descongelar a temperatura ambiente | 2. Centrifugar brevemente | 3. Mezclar a fondo con la pipeta o en vórtex |
|---|---|---|---|
| Rapid Adapter (RA) | No congelado | ✓ | Pipeta |
| Adapter Buffer (ADB) | ✓ | ✓ | Vórtex o pipeta |
| AMPure XP Beads (AXP) | ✓ | ✓ | Mezclar en vórtex justo antes de utilizarlo |
| Elution Buffer (EB) | ✓ | ✓ | Vórtex o pipeta |
| EDTA (EDTA) | ✓ | ✓ | Vórtex o pipeta |
Nota: una vez descongelados, colocar todos los reactivos en hielo.
Añadir 4 μl de EDTA a cada muestra etiquetada con un código de barras, mezclar meticulosamente con la pipeta y centrifugar brevemente.
En este paso se añade EDTA para parar la reacción.
Incubar durante 5 minutos a temperatura ambiente.
Cuantificar 1 μl de cada muestra etiquetada utilizando un fluorímetro Qubit (o equivalente) para realizar el control de calidad.
Agrupar las muestras etiquetadas en proporciones equimolares en un tubo Eppendorf DNA LoBind de 1,5 ml.
Nota: antes de dar este paso, cuantifique las muestras y utilice una concentración equimolar de cada una de ellas, a fin de obtener un balance óptimo de códigos. Tras la PCR, la concentración de las muestras puede variar, por lo que el volumen de cada muestra etiquetada que se añada al conjunto será diferente.
Resuspender las microesferas AMPure XP (AXP) mediante vórtex.
Añadir al conjunto de muestras, un volumen de microesferas AMPure XP resuspendidas, que sea 0,6 veces su volumen y mezclar con la pipeta:
| Volumen del conjunto de muestras etiquetadas | 37,5 μl | 75 μl | 150 μl | 300 μl | 600 μl |
|---|---|---|---|---|---|
| Volumen de AMPure XP Beads (AXP) | 22,5 μl | 45 μl | 90 μl | 180 μl | 360 μl |
Nota: la tabla contiene volúmenes de ejemplo como referencia. Ajustar el volumen añadido de AMPure XP Beads (AXP) al volumen del conjunto de muestras etiquetadas para garantizar una relación de 0,6 veces.
Incubar en el hula mixer (o mezclador rotatorio) durante 5 minutos a temperatura ambiente.
Preparar 2 ml de etanol al 80 % con agua sin nucleasas.
Centrifugar brevemente la muestra y sedimentar en una gradilla magnética hasta que el sobrenadante se vuelva claro e incoloro. Sin mover el tubo, retirar el sobrenadante con una pipeta.
Sin mover el tubo, lavar las microesferas con 1 ml de etanol al 80 %, sin desplazar el sedimento. Retirar el etanol con una pipeta y desechar.
Repetir el paso anterior.
Centrifugar y colocar el tubo de nuevo en la gradilla magnética. Retirar con una pipeta cualquier residuo de etanol. Dejar secar el sedimento durante 30 s aproximadamente, sin dejar que se agriete.
Retirar el tubo de la gradilla y resuspender el sedimento en 15 µl de Elution Buffer (EB). Centrifugar e incubar durante 5 minutos a temperatura ambiente.
Sedimentar las microesferas en una gradilla magnética, durante al menos 1 minuto, hasta que el eluido se vuelva claro e incoloro.
Extraer 15 µl de eluido y guardarlo en un tubo Eppendorf DNA Lobind de 1,5 ml.
- Extraer y colocar el eluido que contiene la biblioteca de ADN, en un tubo de 1,5 ml Eppendorf DNA LoBind.
- Deshechar las microesferas sedimentadas.
Cuantificar 1 μl de muestra eluida utilizando un fluorímetro Qubit.
Transferir 50 fmol de muestra eluida a un tubo nuevo Eppendorf DNA Lobind de 1,5 ml. Añadir Elution Buffer (EB) hasta llegar a un volumen de 11 μl.
En un tubo nuevo Eppendorf DNA LoBind, diluir el adaptador como se indica a continuación y mezclar con la pipeta:
| Reactivo | Volumen |
|---|---|
| Rapid Adapter (RA) | 1,5 μl |
| Adapter Buffer (ADB) | 3,5 μl |
| Total | 5 μl |
Añadir 1 μl de Rapid Adapter (RA) diluido al ADN etiquetado con códigos de barras.
Mezclar dando suaves golpes en el tubo con el dedo y centrifugar brevemente.
Incubar la reacción durante 5 minutos a temperatura ambiente.
La biblioteca preparada se usará para cargar la celda de flujo. Conservar la biblioteca en hielo hasta el momento de cargar.
4. Acondicionar y cargar la celda de flujo MinION/GridION
Material
- Flow Cell Flush (FCF)
- Flow Cell Tether (FCT)
- Library Solution (LIS)
- Library Beads (LIB)
- Sequencing Buffer (SB)
Consumibles
- MinION/GridION Flow Cell
- Seroalbúmina bovina (BSA) (50 mg/ml) (p. ej., Invitrogen™ UltraPure™ BSA 50 mg/ml, AM2616)
- 1.5 ml Eppendorf DNA LoBind tubes
Instrumental
- MinION or GridION device
- MinION/GridION Flow Cell Light Shield
- P1000 pipette and tips
- P100 pipette and tips
- P20 pipette and tips
- P10 pipette and tips
Nótese, este kit es compatible solo con las celdas de flujo R10.4.1 (FLO-MIN114).
Sacar la celda de flujo de la nevera y dejarla a temperatura ambiente durante 20 minutos, mejorará la visibilidad de la matriz durante la preparación y carga de la muestra.
Acondicionar y cargar la celda de flujo
Recomendamos a los usuarios que miren el vídeo Cómo cargar celdas de flujo antes de realizar su primer experimento.
Uso de Library Solution (LIS)
En la mayoría de experimentos de secuenciación, recomendamos usar Library Beads (LIB) para cargar la biblioteca en la celda de flujo. No obstante, podría ser difícil cargar bibliotecas viscosas con las microesferas y tal vez resulte más apropiado utilizar Library Solution (LIS).
Descongelar los viales Sequencing Buffer (SB), Library Beads (LIB) o Library Solution (LIS) —si se requiere— Flow Cell Tether (FCT) y un tubo de Flow Cell Flush (FCF) a temperatura ambiente. Agitar en vórtex. Centrifugar brevemente y poner en hielo.
Añadir seroalbúmina bovina (BSA) a una concentración final de 0,2 mg/ml a la mezcla de preparación de la celda de flujo mejora el rendimiento de secuenciación y la producción de las celdas de flujo MinION R10.4.1 (FLO-MIN114).
Nota: No recomendamos utilizar ningún otro tipo de albúmina (p. ej., seroalbúmina humana recombinante).
Para preparar la mezcla de acondiciomiento con seroalbúmina bovina, combinar los siguientes reactivos en un tubo Eppendorf DNA LoBind de 1,5 ml. Invertir el tubo varias veces y mezclar con la pipeta a temperatura ambiente:
| Reactivos | Volumen por celda de flujo |
|---|---|
| Flow Cell Flush (FCF) | 1 170 µl |
| Seroalbúmina bovina (BSA) a 50 mg/ml | 5 µl |
| Flow Cell Tether (FCT) | 30 µl |
| Volumen total | 1 205 µl |
Abrir la tapa del dispositivo MinION o GridION y deslizar la celda de flujo debajo del clip. Presionar la celda de flujo hacia abajo con firmeza para asegurar un contacto eléctrico y térmico adecuados.

Antes de cargar la biblioteca, verificar la celda de flujo para determinar el número de poros disponible.
Si se ha verificado la celda de flujo con anterioridad, este paso se puede omitir.
Dispone de más información en las instrucciones de comprobación de la celda de flujo, del protocolo de MinKNOW.
Deslizar la tapa del puerto de purgado en el sentido de las agujas del reloj.

Tenga cuidado a la hora de extraer solución amortiguadora de la celda de flujo. No retire más de 20-30 μl y asegúrese de que la solución cubra la matriz de poros en todo momento. La introducción de burbujas de aire en la matriz puede dañar los poros de manera irreversible.
Tras abrir el puerto de purgado, comprobar si hay burbujas de aire bajo la tapa. Retirar una pequeña cantidad de solución amortiguadora para quitar las burbujas:
- Ajustar una pipeta P1000 a 200 μl.
- Introducir la punta de la pipeta en el puerto de purgado.
- Girar la rueda hasta que el indicador de volumen marque 220-230 μl o hasta que se pueda ver una pequeña cantidad de solución amortiguadora entrar en la punta de la pipeta.
Nota: Comprobar que haya un flujo continuo de solución amortiguadora circulando desde el puerto de purgado a través de la matriz de poros.

Cargar 800 μl de mezcla de acondicionamiento por del puerto de purgado, evitando introducir burbujas de aire. Esperar cinco minutos. Durante este tiempo, preparar la biblioteca para cargar siguiendo los pasos a continuación.

Mezclar minuciosamente con la pipeta el contenido del vial Library Beads (LIB).
El vial Library Beads (LIB) contiene microesferas en suspensión. Las microesferas sedimentan muy rápido; por eso, es fundamental mezclarlas justo antes de usar.
En la mayoría de experimentos de secuenciación recomendamos utilizar Library Beads (LIB). El reactivo Library Solution (LIS) está disponible en caso de utilizar bibliotecas más viscosas.
En un tubo nuevo de 1,5 ml Eppendorf DNA LoBind, preparar la biblioteca de la siguiente manera:
| Reactivo | Volumen por celda de flujo |
|---|---|
| Sequencing Buffer (SB) | 37,5 µl |
| Library Beads (LIB) mezcladas justo antes de usar, o Library Solution (LIS), si se requiere | 25,5 µl |
| Biblioteca de ADN | 12 µl |
| Total | 75 µl |
Terminar de acondicionar la celda de flujo:
- Levantar con suavidad la tapa del puerto de carga SpotON.
- Cargar 200 µl de mezcla de acondicionamiento en el puerto de purgado (no en el puerto SpotON), evitando introducir burbujas de aire.

Mezclar la biblioteca suavemente con la pipeta, justo antes de cargar.
Añadir, gota a gota, 75 μl de la biblioteca preparada en el puerto SpotON de la celda de flujo. Procurar que cada gota fluya hacia adentro del puerto antes de añadir la siguiente.

Volver a colocar con cuidado, la tapa del puerto SpotON, procurando que el tapón encaje en el agujero y cerrar el puerto de purgado.


Para obtener resultados de secuenciación óptimos, coloque la pantalla protectora sobre la celda de flujo justo después de cargar la biblioteca.
Recomendamos colocar la pantalla protectora en la celda de flujo y dejarla puesta mientras la biblioteca esté cargada, incluyendo los lavados y pasos de recarga. Retirar la pantalla cuando se haya extraído la biblioteca de la celda de flujo.
Colocar la pantalla protectora de la siguiente manera:
Colocar con cuidado el borde delantero de la pantalla protectora contra el clip. Nota: No hacer fuerza sobre ella.
Posar la pantalla protectora sobre la celda de flujo. La pieza debe asentarse alrededor de la tapa SpotON y cubrir por completo la sección superior de la celda de flujo.

La pantalla protectora no está fijada a la celda de flujo. Una vez colocada, es necesario manipular la celda de flujo con cuidado.
Cerrar la tapa del dispositivo y configurar un experimento de secuenciación en MinKNOW.
Cuando se inserta una celda de flujo en el MinION Mk1D y la tapa del dispositivo se coloca sobre la celda de flujo queda un pequeño espacio alrededor de los lados. Este hecho es normal y no afecta al rendimiento del dispositivo.
Encontrará más información sobre este tema en estas preguntas frecuentes.

5. Adquisición de datos e identificación de bases
Cómo empezar a secuenciar
Una vez la celda de flujo esté cargada, el experimento se pone en marcha desde MinKNOW, el programa de secuenciación que controla el dispositivo, la adquisición de datos y la identificación de bases en tiempo real. Encontrará intrucciones de uso más detalladas en el protocolo de MinKNOW.
Es posible utilizar y configurar MinKNOW para secuenciar de diferentes maneras:
- En un ordenador conectado a un dispositivo de secuenciación, ya sea directamente o a distancia.
- Directamente desde un dispositivo de secuenciación GridION, MinION Mk1C o PromethION 24/48.
Encontrará más información sobre el uso de MinKNOW en los manuales de usuario de los dispositivos:
Cómo empezar un experimento de secuenciación en MinKNOW:
1. Ir a la página principal y pulsar "Iniciar secuenciación".
2. Introducir los datos del experimento, como el nombre, la posición de la celda de flujo y el identificador de la muestra.
3. En la pestaña Kit, seleccionar el kit 16S Barcoding Kit (SQK-16S114-24).
4. En la pestaña Configuración del experimento, ajustar los parámetros de secuenciación y salida del experimento o mantener la configuración por defecto.
Nota: Si la identificación de bases estaba desactivada durante la configuración de un experimento, es posible identificar las bases tras la ejecución en MinKNOW. Para más información, consulte el protocolo de MinKNOW.
5. En la página Inicio, pulsar en la casilla Iniciar la secuenciación.
Análisis de datos
Una vez la secuenciación ha finalizado, es posible reutilizar o devolver la celda de flujo, como se describe en la sección sobre Reutilización o retorno de celdas de flujo.
Tras secuenciar e identificar las bases, es posible analizar los datos. Si desea más información sobre las opciones de análisis disponible durante y tras la identificación de bases, consulte el documento Data Analysis.
En la sección Análisis, se describen otras opciones para analizar los datos.
6. Reutilización y devolución de celdas de flujo
Material
- Kit Flow Cell Wash (EXP-WSH004)
Si al terminar el experimento desea volver a usar la celda de flujo, siga las instrucciones del protocolo Flow Cell Wash Kit y guarde la celda de flujo lavada a entre 2 °C y 8 ⁰C.
El protocolo Flow Cell Wash Kit está disponible en la comunidad Nanopore.
Otra posibilidad es seguir el procedimiento de devolución, lavar la celda de flujo y enviarla a Oxford Nanopore.
Aquí están las instrucciones para devolver celdas de flujo.
Ante cualquier duda o pregunta acerca del experimento de secuenciación, consulte la guía de resolución de problemas, Troubleshooting Guide, que se encuentra en la versión en línea de este protocolo.
7. Análisis posterior
Análisis posterior a la identificación de bases
Recomendamos realizar el análisis utilizando EPI2ME, ya que facilita el análisis bioinformático permitiendo a los usuarios realizar flujos de trabajo Nextflow en una aplicación de escritorio. EPI2ME contiene una colección de flujos de trabajo bioinformáticos, seleccionados y gestionados prontamente por expertos en análisis de secuencias de lecturas largas.
Aquí encontrará más información sobre los flujos de trabajo disponibles en EPI2ME, junto con la Guía de inicio rápido, útiles para abordar el primer flujo de trabajo bioinformático.
El flujo de trabajo 16S (wf-16s) de Nextflow saca el máximo partido de los flujos de trabajo de metagenómica, a fin de identificar el origen de las lecturas a partir de la secuenciación selectiva de amplicones. El flujo de trabajo tiene dos modos de funcionamiento: utiliza kraken2 o minimap2, para determinar el origen de las lecturas.
Aquí encontrará más información sobre el flujo de trabajo 16S de EPI2ME (wf-16s).
Las instrucciones de instalación se encuentran aquí.
Opciones complementarias para análizar más a fondo sus datos:
1. Herramientas para analizar datos de investigación
El departamento de Investigación de Oxford Nanopore Technologies ha creado una serie de herramientas de análisis que están disponibles en el repositorio de GitHub, de Oxford Nanopore. Han sido creadas para usuarios avanzados y contienen instrucciones sobre cómo instalar y ejecutar el programa. Estas herramientas están disponibles públicamente y cuentan con un mantenimiento mínimo.
2. Herramientas de análisis desarrolladas por la comunidad
Si no se proporciona en ninguno de los recursos anteriores un método de análisis que responda a las necesidades de investigación requeridas, consulte la sección Bioinformatics del centro de recursos Resource Centre. Varios miembros de la comunidad Nanopore han desarrollado sus propias herramientas y flujos bioinformáticos para analizar los datos de secuenciación por nanoporos. La mayoría de ellas está disponible en GitHub. Nótese, Oxford Nanopore Technologies no desarrolla ni mantiene esas herramientas y no garantiza que sean compatibles con la última configuración de química o programas informáticos.
8. Problemas durante la extracción de ADN/ARN y la preparación de bibliotecas
A continuación hay una lista de los problemas más frecuentes, con algunas posibles causas y soluciones propuestas.
También disponemos de una página de preguntas frecuentes, FAQ, en la sección Support de la comunidad Nanopore.
Si ha probado las soluciones propuestas y continúa teniendo problemas, póngase en contacto con el departamento de asistencia técnica, bien por correo electrónico (support@nanoporetech.com) o a través del chat Live Support de la comunidad Nanopore.
Baja calidad de la muestra
| Observaciones | Posibles causas | Comentarios y acciones recomendadas |
|---|---|---|
| Baja pureza del ADN (la lectura del Nanodrop para ADN OD 260/280 es <1,8 y OD 260/230 es <2,0-2,2) | El método de extracción de ADN no proporciona la pureza necesaria | Los efectos de los contaminantes se muestran en la página Contaminants. Probar con un método de extracción alternativo que no provoque el arrastre de contaminantes. Considere realizar un paso adicional de limpieza SPRI. |
| Baja integridad del ARN (número de integridad del ARN <9,5 RIN o la banda ARNr se muestra como una mancha en el gel). | El ARN se degradó durante la extracción | Probar un método de extracción de ARN diferente. Encontrará más información sobre el tema en la página RNA Integrity Number. Asimismo, dispone de información adicional en la página DNA/RNA Handling. |
| El ARN tiene una longitud de fragmento más corta de lo esperado | El ARN se degradó durante la extracción | Probar un método de extracción de ARN diferente. Encontrará más información sobre RIN en la página RNA Integrity Number. Asimismo, dispone de información adicional en la página DNA/RNA Handling. Al trabajar con ARN, recomendamos que el espacio de trabajo y el instrumental de laboratorio estén libres de ribonucleasas. |
Escasa recuperación de ADN tras la purificación con microesferas AMPure
| Observaciones | Posibles causas | Comentarios y acciones recomendadas |
|---|---|---|
| Escasa recuperación | Escasa recuperación Pérdida de ADN debido a una proporción de microesferas AMPure por muestra inferior a lo previsto | 1. Las microesferas AMPure sedimentan con rapidez; antes de añadirlas a la muestra hay que asegurarse de que estén bien resuspendidas. 2. Si la proporción de microesferas AMPure por muestra es inferior a 0.4:1, los fragmentos de ADN, sean del tamaño que sean, se perderán durante la limpieza. |
| Escasa recuperación | Los fragmentos de ADN son más cortos de lo esperado | Cuanto menor sea la proporción de microesferas AMPure por muestra, más rigurosa será la selección de fragmentos largos frente a los cortos. Determinar siempre la longitud de la muestra de ADN en un gel de agarosa u otros métodos de electroforesis en gel y, a continuación calcular la cantidad adecuada de microesferas AMPure que se debe utilizar. 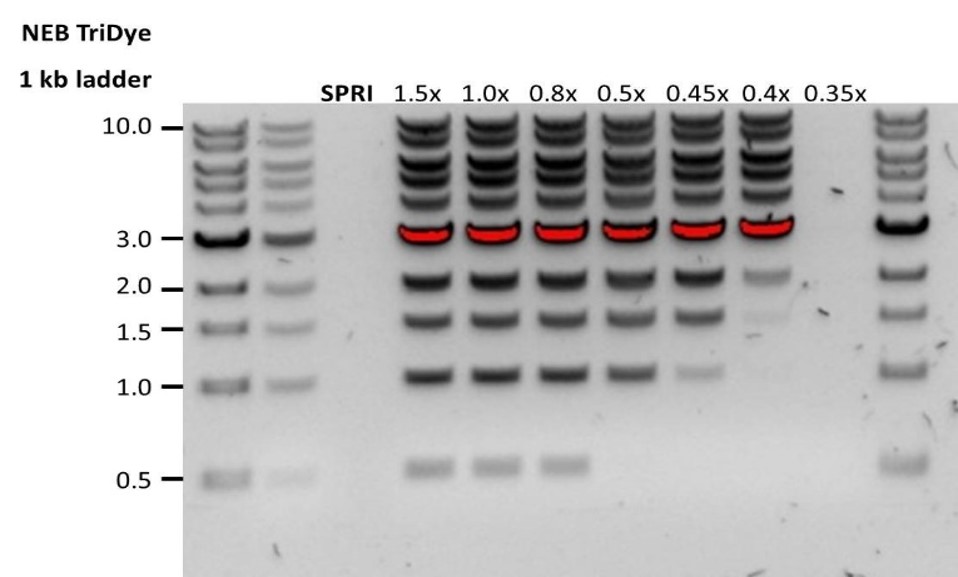 |
| Escasa recuperación tras la preparación de extremos | El paso de lavado utilizó etanol a <80 % | Cuando se utilice etanol a <80 %, el ADN se eluirá de las microesferas. No olvide utilizar el porcentaje correcto. |
9. Problemas durante el experimento al utilizar un kit de secuenciación de base rápida
A continuación hay una lista de los problemas más frecuentes, con algunas posibles causas y soluciones propuestas.
También disponemos de una página de preguntas frecuentes, FAQ, en la sección Support de la comunidad Nanopore.
Si ha probado las soluciones propuestas y continúa teniendo problemas, póngase en contacto con el departamento de asistencia técnica, bien por correo electrónico (support@nanoporetech.com) o a través del chat Live Support de la comunidad Nanopore.
Menos poros al inicio de la secuenciación que tras verificar la celda de flujo
| Observaciones | Posibles causas | Comentarios y acciones recomendadas |
|---|---|---|
| MinKNOW presentó al inicio de la secuenciación un número de poros inferior al indicado durante la comprobación de la celda de flujo | Se introdujo una burbuja de aire en la matriz de nanoporos | Tras comprobar el número de poros presente en la celda de flujo, y antes de acondicionarla, es imprescindible quitar las burbujas que haya cerca del puerto de purgado. Si no se quitan, pueden desplazarse a la matriz de nanoporos y dañar de manera irreversible los nanoporos expuestos al aire. La forma más eficaz de evitar que esto ocurra se muestra en los vídeos Cómo cargar celdas de flujo MinION y Cómo cargar celdas de flujo PromethION. |
| MinKNOW presentó al inicio de la secuenciación un número de poros inferior al indicado durante la comprobación de la celda de flujo | La celda de flujo no está colocada correctamente | Detener el ciclo de secuenciación, quitar la celda de flujo del dispositivo e insertarla de nuevo. Comprobar que está firmemente asentada en el dispositivo y que ha alcanzado la temperatura deseada. Si procede, probar con una posición diferente del dispositivo (GriION/PromethION). |
| MinKNOW presentó al inicio de la secuenciación un número de poros inferior al indicado durante la comprobación de la celda de flujo | La presencia de contaminantes en la biblioteca podría haber dañado o bloqueado los poros | El número de poros resultante tras la evaluación de la celda de flujo se realiza usando el control de calidad de las moléculas de ADN presentes en el tampón de almacenamiento de la celda de flujo. Al inicio de la secuenciación, se utiliza la misma biblioteca para estimar el número de poros activos. Por este motivo, se estima que puede haber una variabilidad del 10 % en el número de poros detectados. Tener un número de poros considerablemente inferior al inicio de la secuenciación podría deberse a la presencia de contaminantes en la biblioteca que hayan dañado las membranas o bloqueado los poros. Para mejorar la pureza del material de entrada tal vez sea necesario usar métodos de purificación o extracción de ADN/ARN alternativos. Los efectos de los contaminantes están descritos en la página Contaminants. Se recomienda, probar con un método de extracción alternativo que no provoque el arrastre de contaminantes. |
Error en el script de MinKNOW
| Observaciones | Posibles causas | Comentarios y acciones recomendadas |
|---|---|---|
| MinKNOW muestra el mensaje "Error en el script" | Reiniciar el ordenador y reiniciar MinKNOW. Si el problema continúa, reúna los archivos de registro MinKNOW log files y contacte con el servicio de asistencia técnica. Si no dispone de otro dispositivo de secuenciación, recomendamos que guarde la celda de flujo cargada a 4 °C y contacte con el servicio de asistencia técnica para recibir instrucciones de almacenamiento adicionales. |
Ocupación de poro por debajo del 40 %
| Observaciones | Posibles causas | Comentarios y acciones recomendadas |
|---|---|---|
| Ocupación del poro < al 40 % | No se cargó suficiente biblioteca en la celda de flujo | En una celda de flujo MinION o GridION se cargan de 10 a 50 fmol de biblioteca de buena calidad. Cuantificar la biblioteca antes de cargarla y calcular moles con herramientas como la calculadora Biomath de Promega, (opción "dsDNA: μg to pmol"). |
| Ocupación de los poros próxima a 0 | Se utilizó el kit, Rapid PCR Barcoding V14 y los adaptadores de secuenciación no se ligaron al ADN. | No olvidar seguir el protocolo paso a paso y utilizar los volúmenes y las temperaturas de incubación correctos. Es posible preparar una biblioteca de lambda control para valorar la integridad de los reactivos. |
| Ocupación de los poros próxima a 0 | No hay anclaje en la celda de flujo | Los anclajes se añaden mientras se acondiciona la celda de flujo (vial FCT). El anclaje debe añadirse al enjuague (vial FCF) antes de acondicionar la celda de flujo. |
Longitud de lectura más corta de lo esperado
| Observaciones | Posibles causas | Comentarios y acciones recomendadas |
|---|---|---|
| Longitud de lectura más corta de lo esperado | Fragmentación no deseada de la muestra de ADN | La longitud de lectura refleja la longitud del fragmento de la muestra de ADN. La muestra de ADN se puede fragmentar durante la extracción y preparación de la biblioteca. 1. Consulte la sección de buenas prácticas de extracción en la página Extraction Methods de la comunidad Nanopore. 2. Visualizar la distribución de la longitud de los fragmentos de las muestras de ADN en un gel de agarosa antes de proceder a la preparación de la biblioteca. 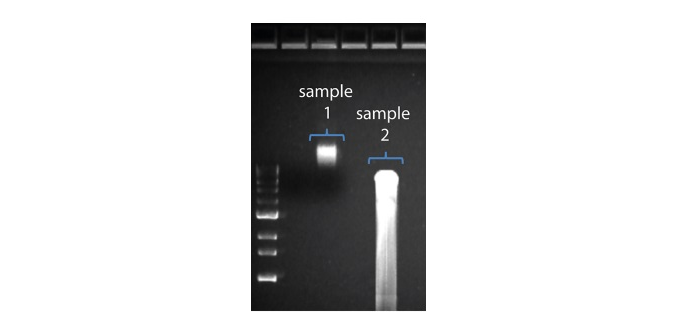 En la imagen superior, la muestra 1 contiene alto peso molecular, mientras que la muestra 2 se ha fragmentado. En la imagen superior, la muestra 1 contiene alto peso molecular, mientras que la muestra 2 se ha fragmentado.3. Durante la preparación de la biblioteca, evitar pipetear y agitar en vórtex cuando se mezclen los reactivos. Dar suaves golpes con el dedo o invertir el vial es suficiente. |
Gran proporción de poros no disponibles
| Observaciones | Posibles causas | Comentarios y acciones recomendadas |
|---|---|---|
Gran proporción de poros no disponibles (se muestran en azul en el panel de canales y en el gráfico de actividad de poros) 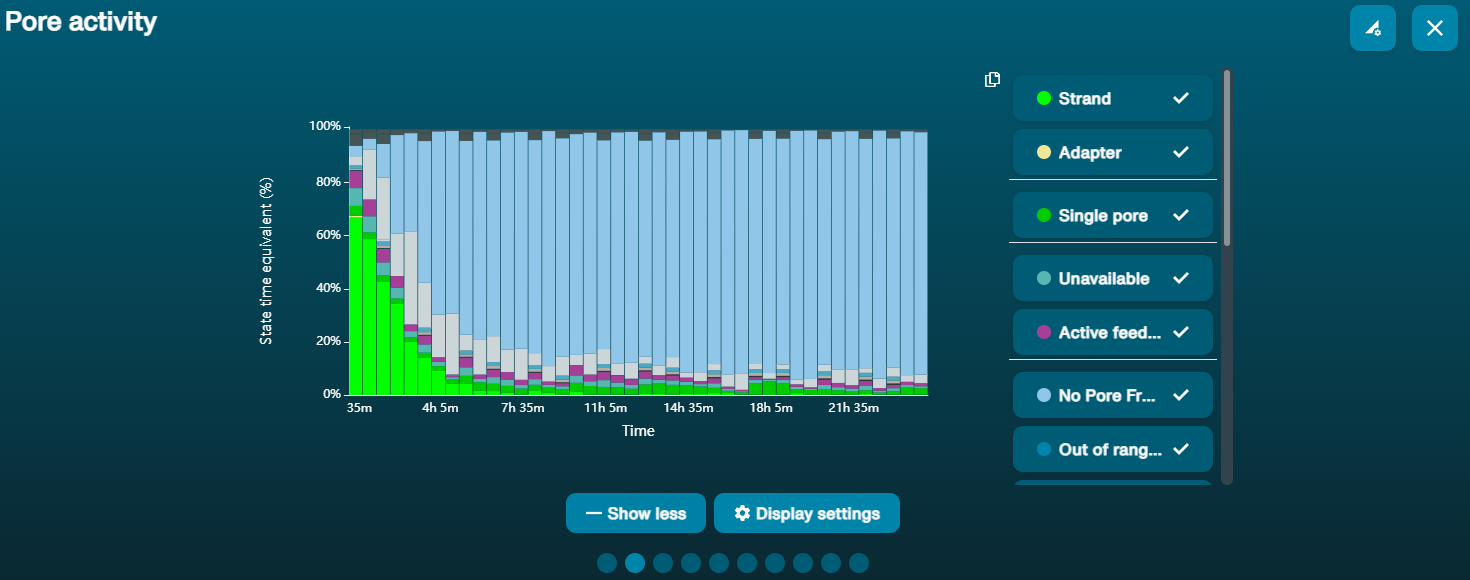 Conforme pasa el tiempo, el gráfico de actividad de poros de arriba muestra una proporción creciente de poros "no disponibles". Conforme pasa el tiempo, el gráfico de actividad de poros de arriba muestra una proporción creciente de poros "no disponibles". | Hay contaminantes presentes en la muestra | Algunos contaminantes se pueden eliminar de los poros mediante la función de desbloqueo incorporada en MinKNOW. Si funciona, el estado de los poros cambiará a "sequencing pores". Si la porción poros no disponibles se mantiene elevada o aumenta: 1. Realizar un purgado de nucleasa con el kit de lavado Flow Cell Wash Kit (EXP-WSH004) 2. Realizar varios ciclos de PCR para intentar diluir cualquier contaminante que pueda estar causando problemas. |
Gran proporción de poros inactivos
| Observaciones | Posibles causas | Comentarios y acciones recomendadas |
|---|---|---|
| Gran proporción de poros inactivos/no disponibles (se muestran en azul claro en el panel de canales y en el gráfico de actividad de poros. Los poros o membranas están dañados de manera irreversible) | Se han introducido burbujas de aire en la celda de flujo | Las burbujas de aire introducidas durante el acondicionamiento de la celda y carga de la biblioteca podrían dañar los poros de forma permanente. La forma más eficaz de evitar que esto ocurra se muestra en los vídeos Cómo cargar celdas de flujo MinION y Cómo cargar celdas de flujo PromethION. |
| Gran proporción de poros inactivos/no disponibles | Ciertos compuestos copurificados con ADN | Compuestos conocidos, incluidos los polisacáridos, se asocian generalmente con el ADN genómico de las plantas. 1. Consulte los métodos de extracción de ADN en la página Plant leaf DNA extraction method. 2. Purificar con el kit QIAGEN PowerClean Pro. 3. Realizar una amplificación del genoma completo con la muestra original de ADNg utilizando el kit QIAGEN REPLI-g. |
| Gran proporción de poros inactivos/no disponibles | Hay contaminantes presentes en la muestra | Los efectos de los contaminantes se muestran en la página Contaminants. Probar con un método de extracción alternativo que no provoque el arrastre de contaminantes. |
Fluctuación de la temperatura
| Observaciones | Posibles causas | Comentarios y acciones recomendadas |
|---|---|---|
| Fluctuación de la temperatura | La celda de flujo ha perdido contacto con el dispositivo | Comprobar que una almohadilla térmica cubra la placa metálica de la parte posterior de la celda de flujo. Reinsertar la celda de flujo y presionar para asegurarse de que las clavijas del conector estén bien conectadas al dispositivo. Si el problema continúa, contacte con el servicio de asistencia técnica. |
Error al intentar alcanzar la temperatura deseada
| Observaciones | Posibles causas | Comentarios y acciones recomendadas |
|---|---|---|
| MinKNOW muestra el mensaje "Error al intentar alcanzar la temperatura deseada" | El dispositivo ha sido colocado en un lugar con una temperatura ambiente inferior a la media o en un lugar con escasa ventilación (lo que provoca el sobrecalientamiento de las celdas de flujo). | MinKNOW dispone de un tiempo predeterminado para que las celdas de flujo alcancen la temperatura fijada. Una vez transcurrido ese tiempo, aparece un mensaje de error, pero el experimento de secuenciación continúa. Secuenciar a una temperatura incorrecta puede provocar disminuciones en el rendimiento y generar índices de calidad Qscore menores. Corrija la ubicación del dispositivo, procurando que esté a temperatura ambiente y tenga buena ventilación; a continuación, reinicie el proceso en MinKNOW. Encontrará más información sobre el control de temperatura del MinION en este enlace. |
10. Diseño de cebadores, consideraciones sobre la PCR y la secuenciación
Nuestros conjuntos de cebadores están alineados con los estándares de la industria y proporcionan cobertura de todo el gen 16S ARNr. Sin embargo, los cebadores utilizados se unen a regiones que no están conservadas al 100 % entre especies, por lo tanto no podemos garantizar la amplificación en todas ellas.
Haga un experimento de control y compruebe si la especie de interés se amplifica con los cebadores proporcionados en este kit. Si necesita asistencia adicional, escríbanos a support@nanoporetech.com.
Diseño de cebadores 16S
Los cebadores 16S suministrados con este kit no han mostrado ningún indicio que propicie la dimerización ni la formación de horquillas.







